Γιατί τα Άκρα του Γονιδιώματος Είναι Σημαντικά
Ανακαλύπτοντας τι μπορούμε να μάθουμε από την προσομοίωση της αλληλουχίας shotgun
Μετάφραση: Απολλόδωρος
14 Ιουλίου 2023 | USMORTALITY | Διαβάστε το εδώ
Εισαγωγή
Πριν από δύο εβδομάδες, αφού δημοσίευσα άλλη μια συνέχεια της σειράς μου για το γονιδίωμα, μπήκα σε μια διαφωνία με ανθρώπους στο Twitter. Πριν δημοσιεύσω μια λεπτομερή απάντηση, θέλω πρώτα να εξηγήσω λεπτομερώς, γιατί κατά τη γνώμη μου, είναι σημαντικό να κατανοήσουμε γιατί η εξέταση των άκρων του γονιδιώματος, κατά τη γνώμη μου, είναι τόσο σημαντική.
Όπως περιγράφηκε προηγουμένως, το αρχικό δείγμα του ασθενούς περιείχε πολλές διαφορετικές πηγές RNA (ανθρώπινο, άλλων ειδών, ιογενές). Ως εκ τούτου, εάν οι συγγραφείς των Wu et al. 2020, υπέθεσαν ότι υπήρχε ένας νέος ιός (ή καλύτερα πολλαπλοί ιοί) στο δείγμα, τότε το πλήρες γονιδίωμα θα έπρεπε να βρίσκεται στο δείγμα.
Επισκόπηση της γονιδιωματικής σειράς
Κατακερματισμός του RNA
Ένα βήμα στη διαδικασία εξαγωγής RNA, όπως περιγράφεται από τους Wu et al. 2020, εξηγεί τη χρήση του "SMARTer Stranded Total RNA-Seq kit v.2" της Takara, Inc.
Ένα βήμα αυτού του πρωτοκόλλου είναι η διάσπαση του ολικού RNA σε μικρά κομμάτια για το βήμα της αλληλούχισης, η οποία μπορεί να γίνει μέσω θερμότητας ή ενζύμων.
Επικοινώνησα με την Τεχνική Υποστήριξη της Takara, προκειμένου να ρωτήσω "αν η θέση της διάσπασης του RNA κατά το βήμα του τεμαχισμού είναι τυχαία ή προκαθορισμένη;". - Ακολουθεί η απάντησή τους:
Σύμφωνα λοιπόν με αυτήν, η διαδικασία οδηγεί σε τυχαία κομμάτια RNA. Με βάση αυτό, μπορούμε τώρα να προσομοιώσουμε αυτή τη διαδικασία:
Να χρησιμοποιήσουμε ή να δημιουργήσουμε ένα γονιδίωμα αναφοράς
Κατακερματισμός σε τυχαία κομμάτια/αναγνώσεις μήκους ~100bp
Φίλτρο αναγνώσεων μήκους 50-150bp
Ευθυγράμμιση (ή de-novo assemble) των αναγνωσμάτων που προκύπτουν με το γονιδίωμα αναφοράς.
Προσομοιωμένα αποτελέσματα
Έχω γράψει ένα σενάριο για την προσομοίωση της παραπάνω διαδικασίας χρησιμοποιώντας το γονιδίωμα αναφοράς SARS-CoV-2 (MN908947.3).
Το σενάριό μου ρυθμίστηκε έτσι ώστε να ξεκινάει με 100 άθικτα γονιδιώματα και ποσοστό σφάλματος ανάγνωσης 1%. Ακολουθούν τα αποτελέσματα ευθυγράμμισης αυτών των τυχαία παραγόμενων αναγνώσεων (με bwa mem):
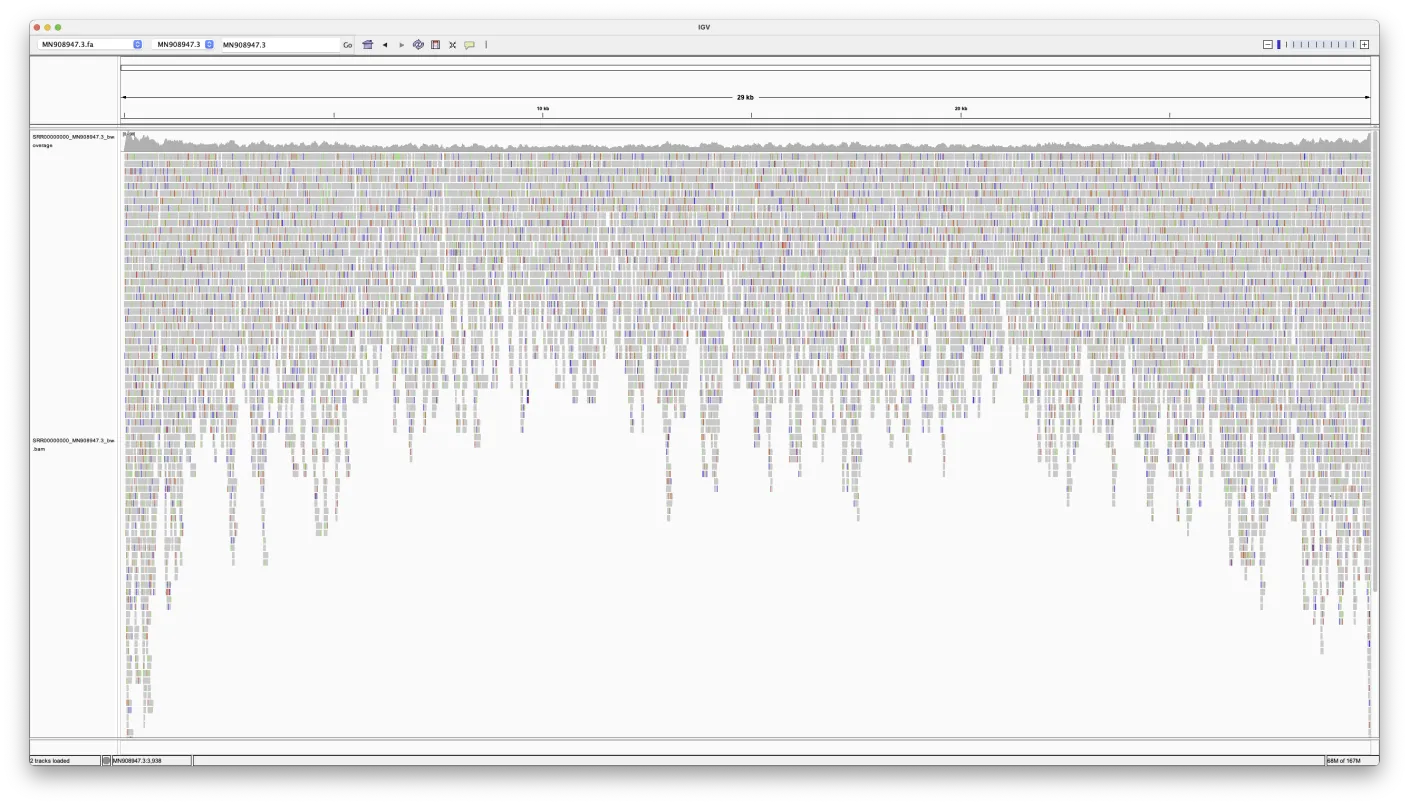
Ολόκληρο γονιδίωμα
Σημειώστε, πώς τα reads συσσωρεύονται προς τα άκρα.
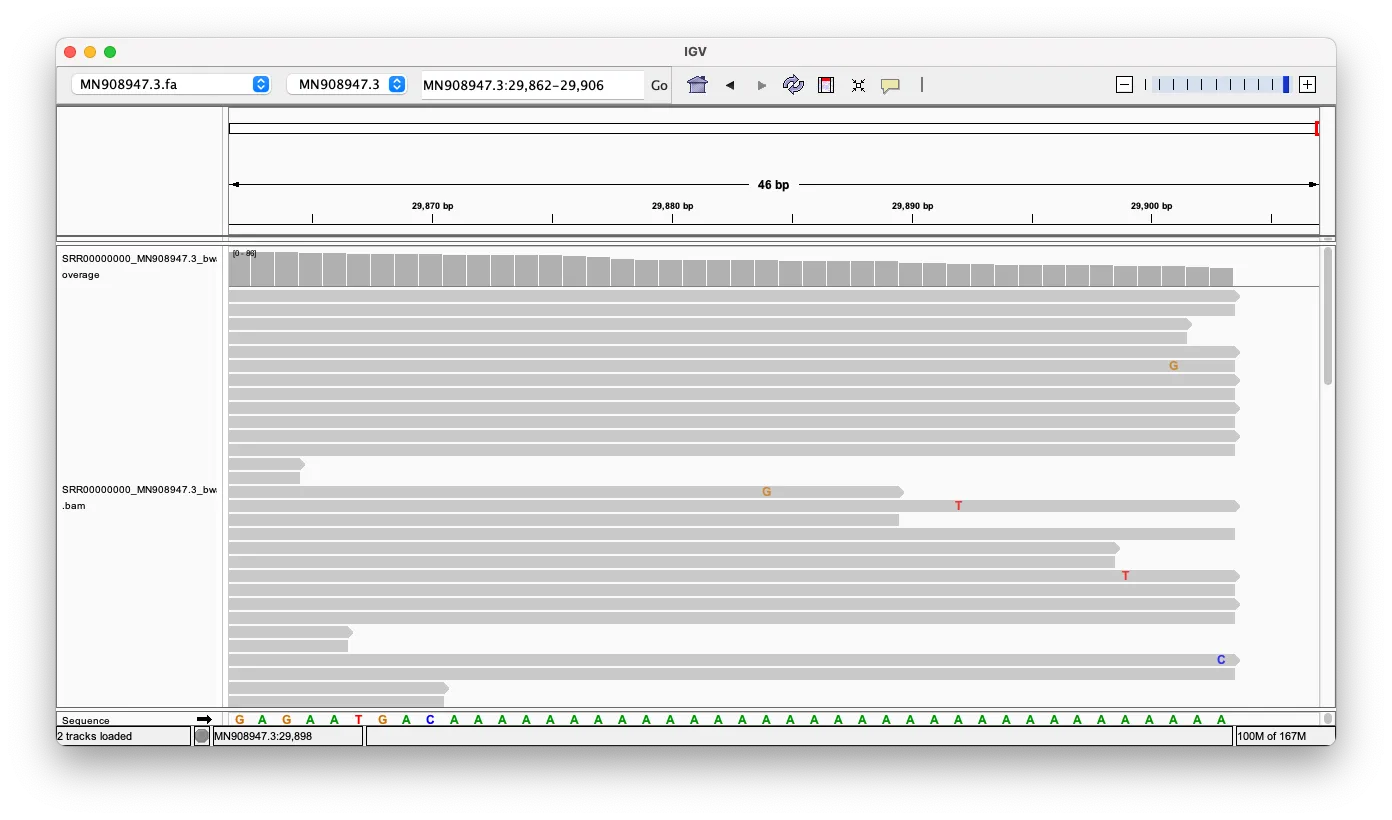
Κεφαλή
Κάνοντας ζουμ στην περιοχή της κεφαλής του γονιδιώματος, μπορούμε να δούμε ότι υπάρχουν 16 αναγνώσματα που ευθυγραμμίζονται τέλεια με την κεφαλή. Δεν υπάρχουν τυχαίες προσθήκες ή διαγραφές, αλλά ούτε και μαζικά σφάλματα ανάγνωσης.
Ουρά
Ίδια εικόνα εδώ, βλέπουμε (τουλάχιστον) 14 αναγνώσματα που ευθυγραμμίζονται τέλεια με την ουρά.
Επανέλαβα αυτό το βήμα 20x και με 100 ιούς και 1% ποσοστό σφάλματος ανάγνωσης το μέσο βάθος ήταν 24 αναγνώσεις με τυπική απόκλιση 6.
Συναρμολόγηση De-Novo
Η χρήση του Megahit για την de-novo συναρμολόγηση αυτών των αναγνώσεων, οδηγεί σταθερά σε (σχεδόν) ολόκληρο το γονιδίωμα, εάν το βάθος (πόσα γονιδιώματα αναφοράς χρησιμοποιούνται) είναι επαρκές. Σε αυτή την περίπτωση, με n=100, έχουμε μια σχεδόν τέλεια συναρμολόγηση:
1 contigs, συνολικά 29890 bp, min 29890 bp, max 29890 bp, avg 29890 bp, N50 29890 bp
Σε αυτή την περίπτωση συναρμολογήθηκε το 29890/29903 = 99,99% του γονιδιώματος και βρέθηκε μόνο ένα contig, κυρίως επειδή δεν υπήρχε άλλο RNA στο δείγμα.
Οι 13 βάσεις που έλειπαν έλειπαν από την ουρά polyA, ενώ η κεφαλή ανακατασκευάστηκε τέλεια. Η επανάληψη της διαδικασίας με n=1.000 είχε ως αποτέλεσμα να λείπουν 6 βάσεις στο τέλος, αλλά ακόμη και με n=10.000 το Megahit δεν μπόρεσε να συναρμολογήσει πλήρως το τέλος.
Θα εμβαθύνω σε αυτό με περισσότερες λεπτομέρειες με το επόμενο μέρος της σειράς.
Περίληψη
Έτσι, συνοψίζοντας, μπορούμε να δούμε ότι χρησιμοποιώντας 100 γονιδιώματα αναφοράς και ένα ποσοστό σφάλματος 1%, καταλήγουμε σε περίπου ~15 τέλεια ευθυγραμμισμένες αναγνώσεις κατά την κεφαλή και την ουρά (και μια γενική κάλυψη 24). Αυτό σημαίνει ότι αν ένα ολόκληρο γονιδίωμα υπήρχε στο δείγμα, θα πρέπει να είμαστε σε θέση να βρούμε αναγνώσματα που ευθυγραμμίζονται τέλεια έναντι αυτού του νέου γονιδιώματος.
Το Megahit συναρμολόγησε πλήρως την κεφαλή και το "σώμα" του γονιδιώματος, αλλά δυσκολεύτηκε με την (polyA)-ουρά. Αυτό θα μπορούσε να οφείλεται στο μέσο μήκος ανάγνωσης των 100bp, αλλά η ουρά έχει μόνο 34x A, οπότε παραμένει ασαφές γιατί το Megahit δεν είναι σε θέση να συναρμολογήσει πλήρως το γονιδίωμα.
Δικτυογραφία:
Why the Ends of the Genome are Important - USMortality



![Είναι Έγκυρο το Γονιδίωμα του SARS-CoV-2; -- [Μέρος 3ο]](https://substackcdn.com/image/fetch/$s_!5hOo!,w_140,h_140,c_fill,f_auto,q_auto:good,fl_progressive:steep,g_auto/https%3A%2F%2Fsubstack-post-media.s3.amazonaws.com%2Fpublic%2Fimages%2F45b877b3-d2d1-4c9c-9724-dc083620532e_1200x600.png)
![Συναρμολόγηση του Γονιδιώματος (Genome Assembly) του SARS-CoV-2 -- [Μέρος 2ο]](https://substackcdn.com/image/fetch/$s_!7Vk6!,w_140,h_140,c_fill,f_auto,q_auto:good,fl_progressive:steep,g_auto/https%3A%2F%2Fsubstack-post-media.s3.amazonaws.com%2Fpublic%2Fimages%2F51bea0ea-4454-4f70-8b11-0e354dc27269_643x397.png)
![Συναρμολόγηση του Γονιδιώματος (Genome Assembly) του SARS-CoV-2 -- [Μέρος 1ο]](https://substackcdn.com/image/fetch/$s_!GImO!,w_140,h_140,c_fill,f_auto,q_auto:good,fl_progressive:steep,g_auto/https%3A%2F%2Fsubstack-post-media.s3.amazonaws.com%2Fpublic%2Fimages%2F09c39106-a905-47a6-a089-3162b3300890_1075x600.png)