Είναι Έγκυρο το Γονιδίωμα του SARS-CoV-2; -- [Μέρος 3ο]
Μια πιο προσεκτική ματιά στη χαρτογράφηση των πρώτων γενετικών αναγνώσεων.
Μετάφραση: Απολλόδωρος
28 Ιουνίου 2023 | USMortality | Διαβάστε το εδώ
Η ευθυγράμμιση των αρχικών αναγνώσεων που δημοσιεύονται από τους Wu et al. 2020 με το γονιδίωμα αναφοράς SARS-CoV-2 αποδίδει κάλυψη 99,993%. Ωστόσο, μια πιο προσεκτική εξέταση των ευθυγραμμίσεων, ειδικά για την κεφαλή και την ουρά του νέου γονιδιώματος, αποκαλύπτει ότι καμμία από τις αρχικές αναγνώσεις δεν ταιριάζει επαρκώς ώστε να δημιουργηθεί βεβαιότητα για την εγκυρότητα του γονιδιώματος αναφοράς.
Θα πρέπει τουλάχιστον να αποδειχθεί ότι:
Τουλάχιστον 10-20 αναγνώσεις (reads) που παρουσιάζουν τέλεια στοίχιση με την κεφαλή & την ουρά.
Στο δείγμα υπάρχει πλήρης γενετική αλληλουχία (RNA) μήκους ~30kb.
Εξ όσων γνωρίζω, καμμία από αυτές τις δύο προϋποθέσεις δεν έχει εκπληρωθεί μέχρι σήμερα.
Ιστορικό
Οι Wu et al., 2020 ήταν η πρώτη μελέτη που δημοσίευσε την αρχική αλληλουχία. Στην πρώτη μου δημοσίευση για το θέμα αυτό, εξήγησα λεπτομερώς πώς εργάστηκαν οι συγγραφείς για την ανάκτηση της πρώτης αλληλουχίας. Σε μια επόμενη ανάρτηση, συζήτησα περισσότερες ανωμαλίες & ανοιχτά ερωτήματα γύρω από τα ευρήματα του ισχυριζόμενου ιικού γονιδιώματος του SARS-CoV-2, του υποτιθέμενου παθογόνου που προκαλεί το COVID-19. Τα βασικά συμπεράσματα ήταν, ότι υπήρχαν σημαντικά προβλήματα στην αναπαραγωγή του πλήρους γονιδιώματος μέσω της de-novo συναρμολόγησης Megahit, συγκεκριμένα, ότι:
Το μεγαλύτερο υποτιθέμενο μήκος contig, το οποίο χρησιμοποιήθηκε ως βάση για το γονιδίωμα, μέχρι σήμερα, δεν έχει αναπαραχθεί πανομοιότυπα από κανέναν, απ' όσο γνωρίζω.
Η ακριβής αρχή και η πλήρης ουρά του γονιδιώματος δεν κατασκευάστηκαν από το Megahit, τον συναρμολογητή που παρήγαγε το μακρύτερο αρχικό contig.
Πολλές μελέτες τώρα, δεν χρησιμοποιούν πλέον de-novo assembly, αλλά αντ' αυτού απλώς αντιστοιχίζουν τα short-reads στο "γονιδίωμα αναφοράς". Ακολουθεί μια γρήγορη επισκόπηση της ροής εργασίας:
Εισαγωγή
Ενώ η θεωρητική συναρμολόγηση μέσω του αλγορίθμου γραφήματος De Bruijn (επεξηγηματικό βίντεο εδώ) φαίνεται να ισχύει - για ένα νέο γονιδίωμα, τα άκρα θα πρέπει να είναι ιδιαίτερα σημαντικά. Κάποιος θα μπορούσε σίγουρα να περιμένει, να βρει αναγνώσεις, που αντιστοιχούν τέλεια στην κεφαλή (και την ουρά) του γονιδιώματος - φανταστείτε το παρόμοιο με ένα κομμάτι ακμής ενός παζλ!
Ως εκ τούτου, αν εξετάσουμε τις μεμονωμένες αναγνώσεις, θα περίμενα να βρω πολλές αναγνώσεις, οι οποίες ευθυγραμμίζονται τέλεια με τα άκρα του γονιδιώματος αναφοράς (MN908947.3).
Μέθοδοι
Για τη στοίχιση των αναγνώσεων, έτρεξα μια χούφτα κοινές εντολές βιοπληροφορικής, οι οποίες συνοψίζονται σε αυτό το σενάριο: ./align.sh SRR10971381 MN908947.3
Στη συνέχεια χρησιμοποίησα το Integrative Genomics Viewer (IGV) για την οπτικοποίηση.
Αποτελέσματα
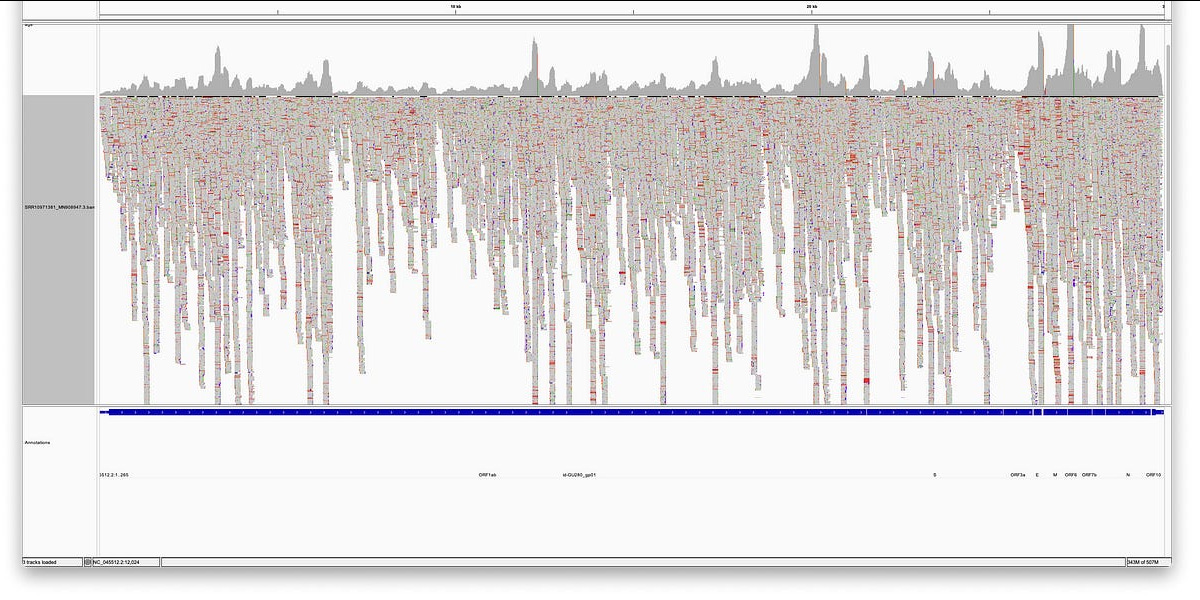
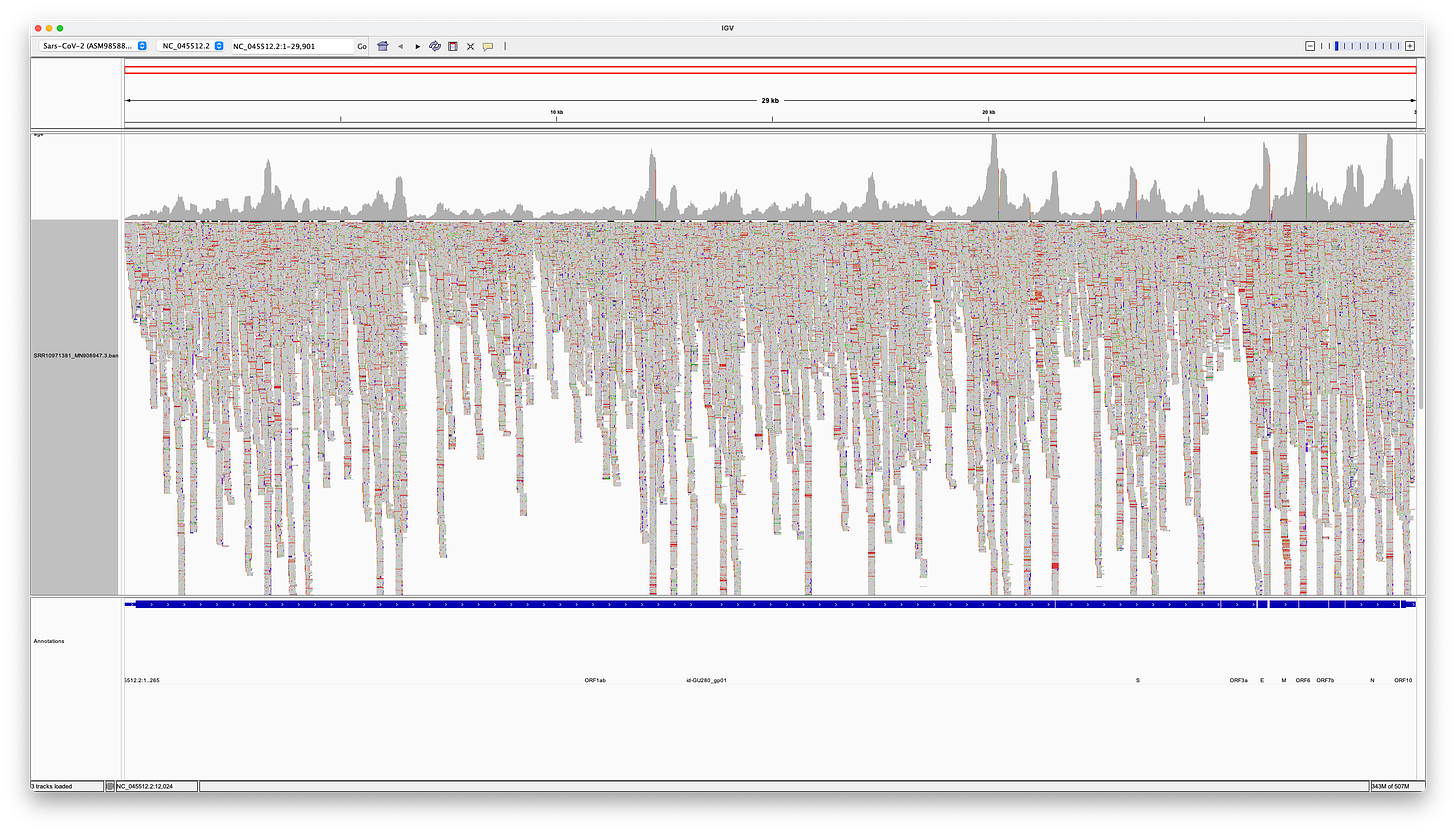
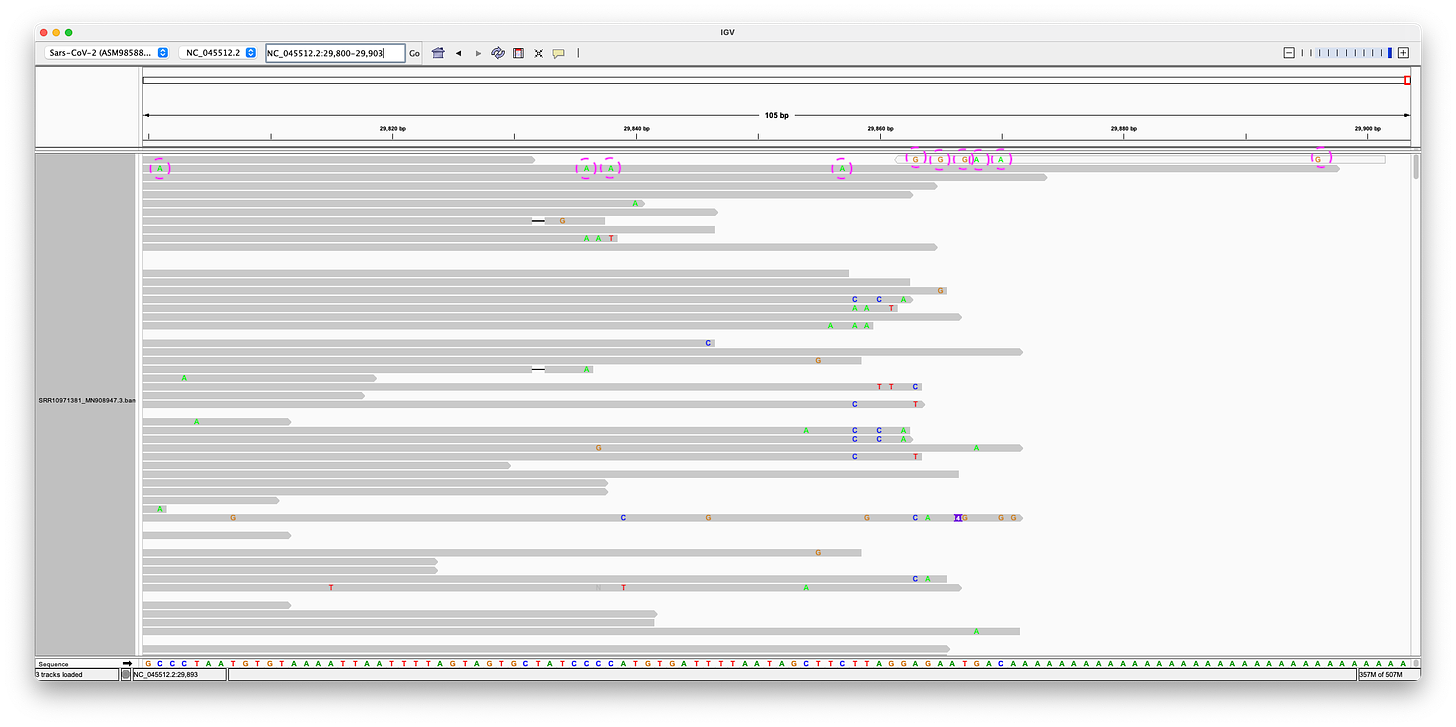
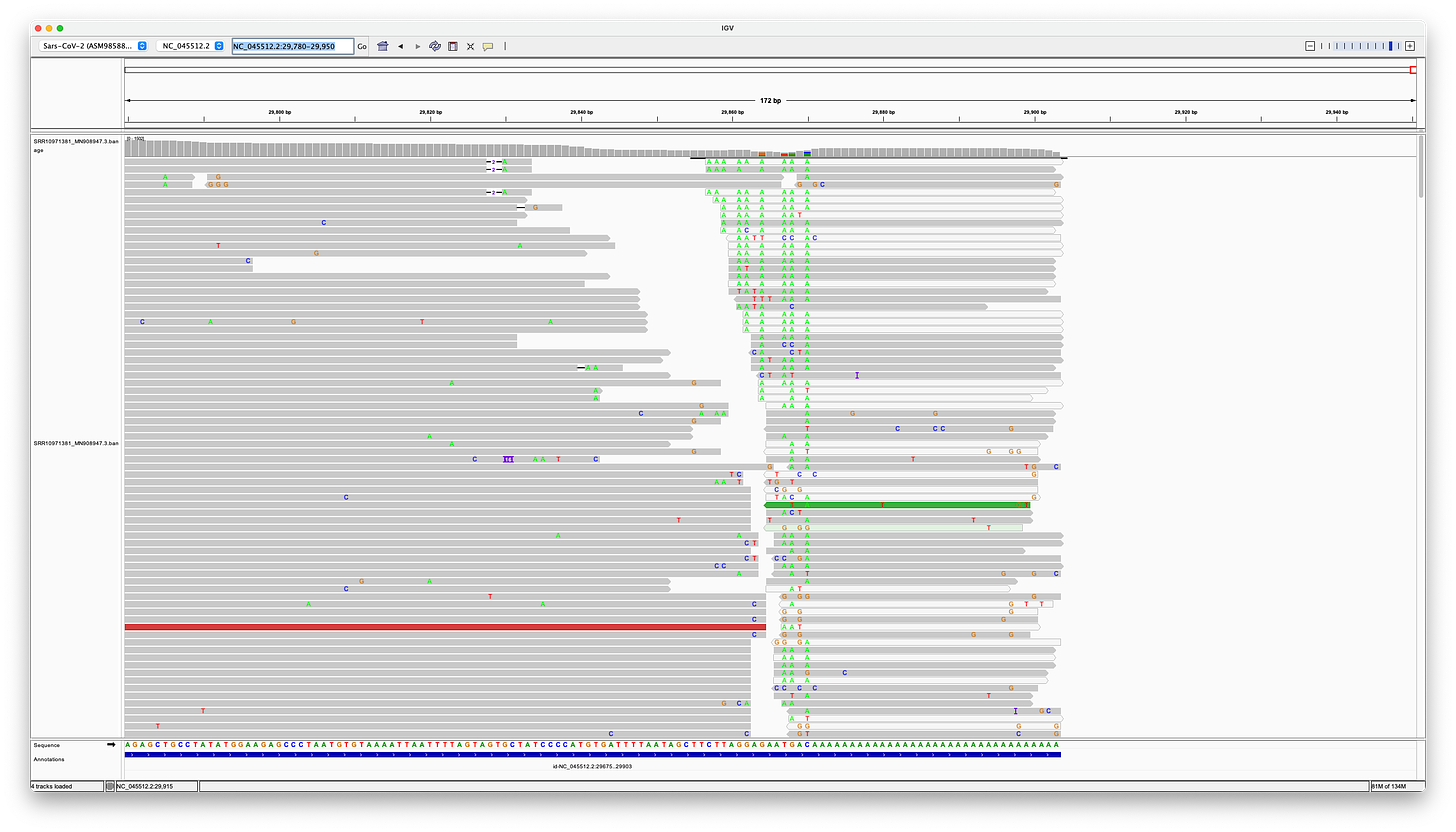
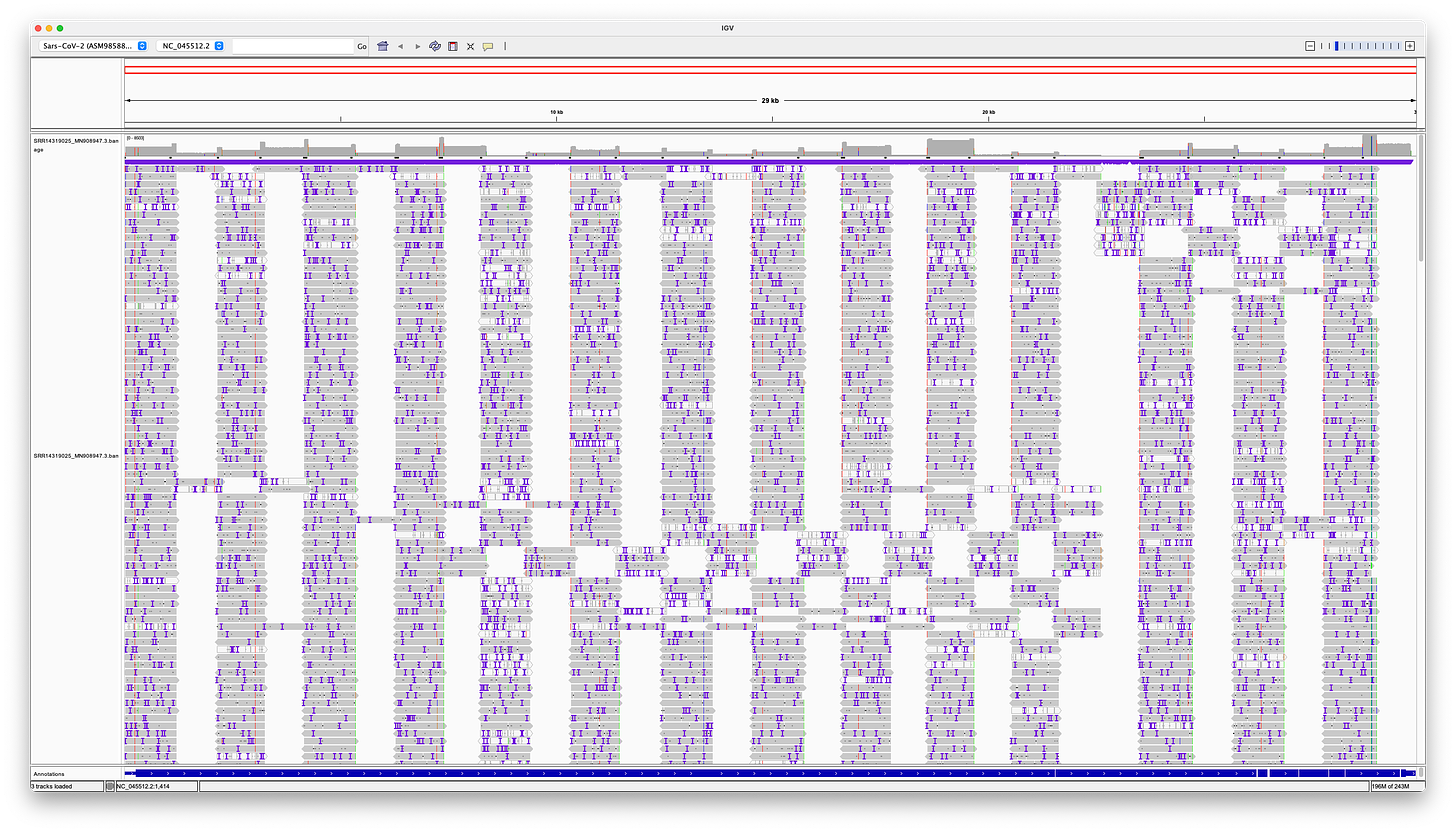
Ιδού λοιπόν τι δείχνει το IGV: Η πρώτη σειρά δείχνει την κάλυψη, η δεύτερη όλες τις αναγνώσεις, οι οποίες ευθυγραμμίζονται με το γονιδίωμα αναφοράς (SARS-CoV-2 v3) στην τρίτη σειρά (μπλε).
Μπορεί ήδη να δει κανείς, ότι είναι ορατός ένας πολύς τονισμένος χρωματισμός, ο οποίος αναδεικνύει αναντίστοιχες βάσεις, ένθετα και διαγραφές, και ότι η κάλυψη ποικίλλει σε μεγάλο βαθμό.
Κεφαλή
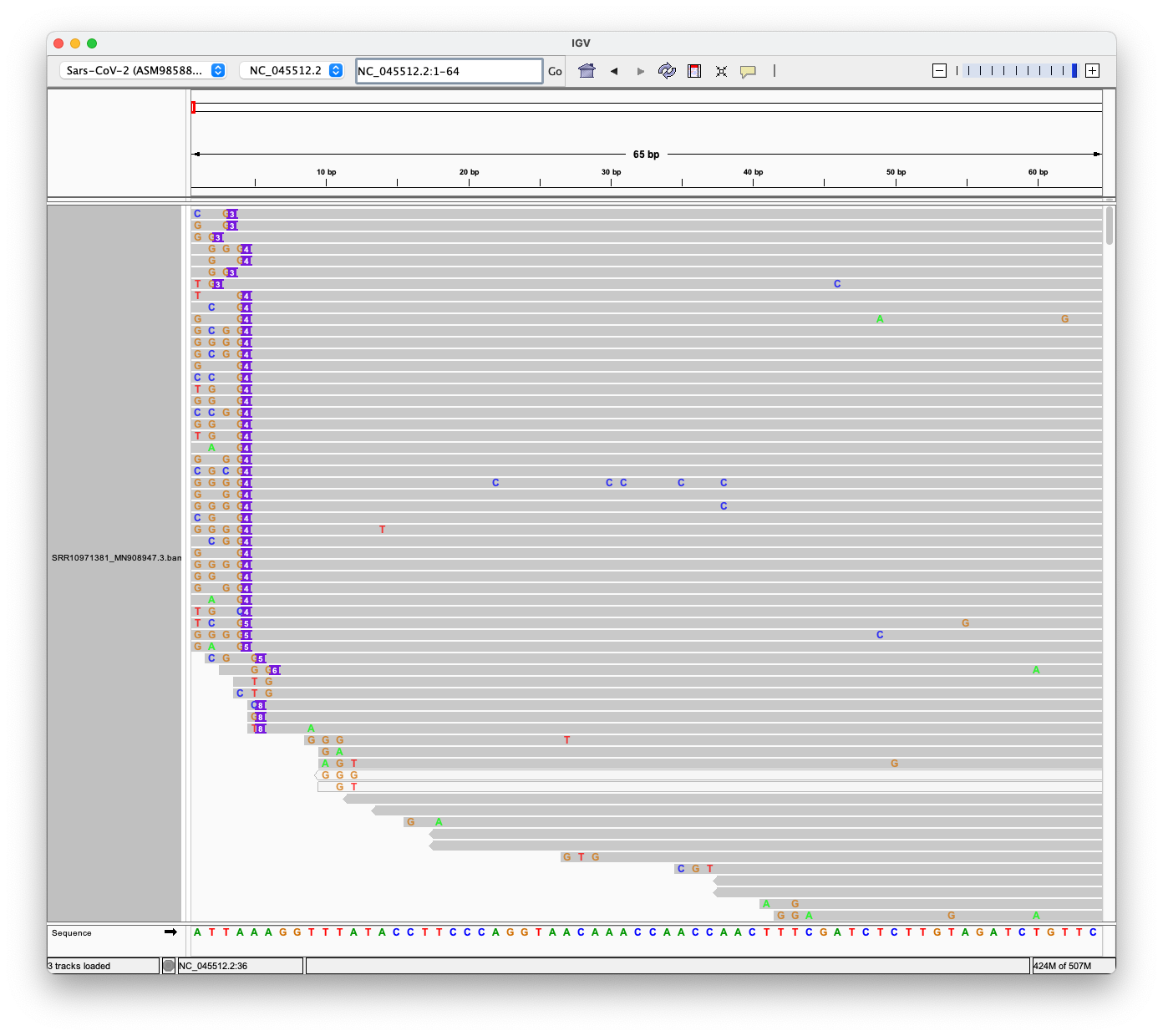
Ας δούμε το "αριστερό χέρι/αρχή" της αρχικής αλληλουχίας των Fan Wu et al.
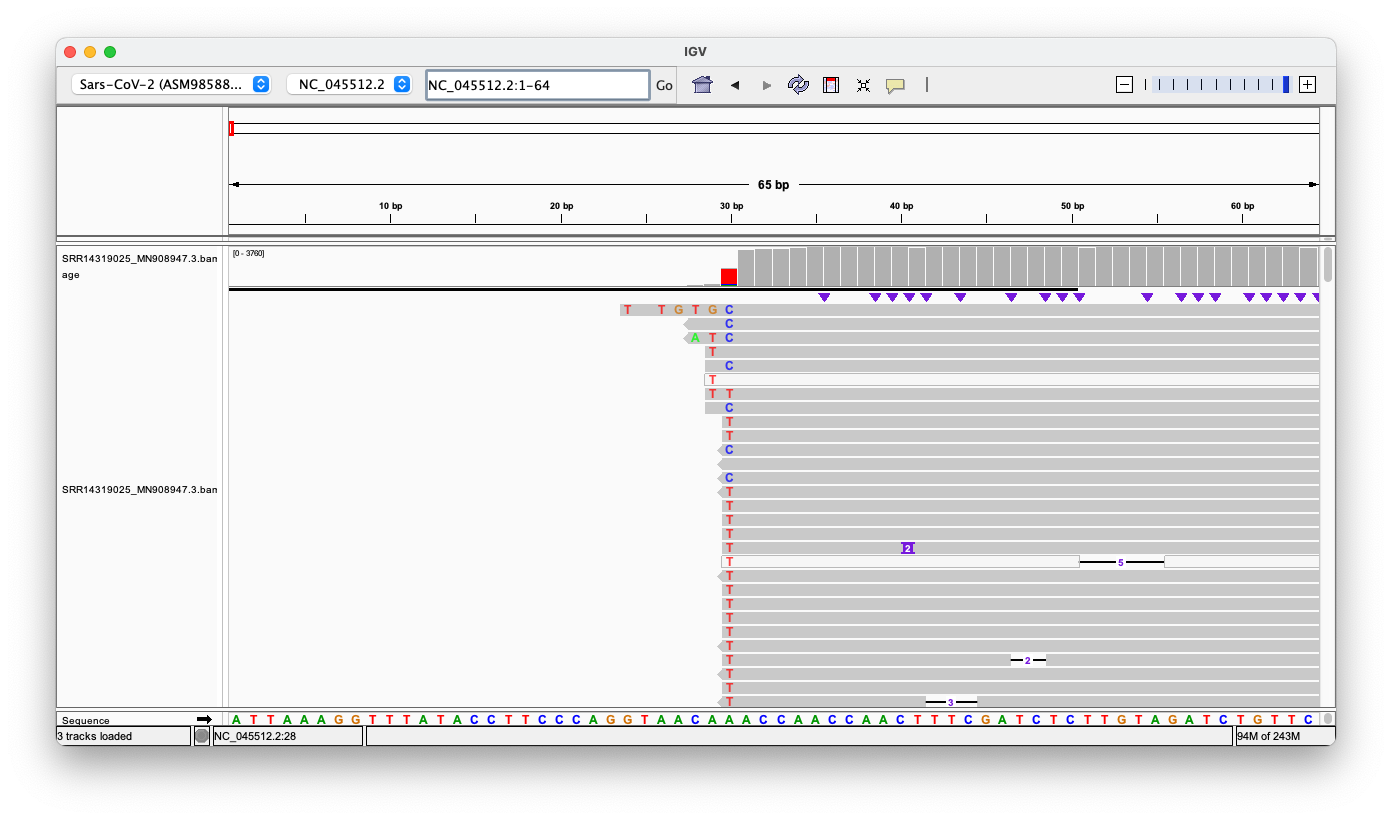
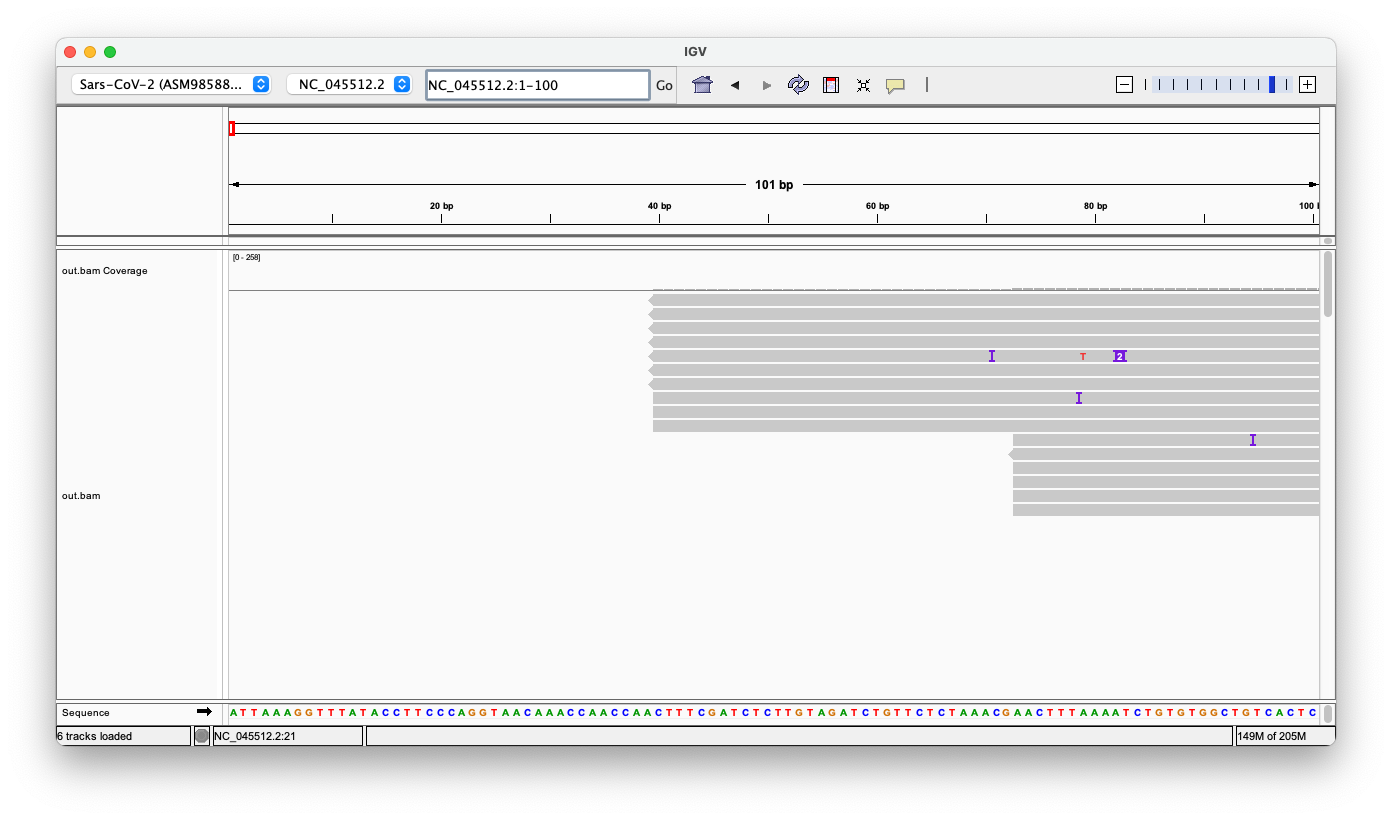
Εδώ είναι οι πρώτες 64 βάσεις. Στο κάτω μέρος είναι η επίσημη αλληλουχία αναφοράς. Μόνο 28 αναγνώσεις ευθυγραμμίζονται από την αρχή, επτά επιπλέον αναγνώσεις ευθυγραμμίζονται με λιγότερες από τέσσερις βάσεις να λείπουν.
Ωστόσο, όλες οι αναγνώσεις έχουν ένθετα ή αναντιστοιχίες - ούτε μία ανάγνωση δεν ταιριάζει απόλυτα!
Για ένα νέο γονιδίωμα, θα περίμενε κανείς σίγουρα, να βρει αναγνώσματα, που αντιστοιχούν τέλεια στην αρχή του γονιδιώματος - φανταστείτε το παρόμοιο με ένα κομμάτι άκρης ενός παζλ!
Τι είναι οι αναντιστοιχίες;
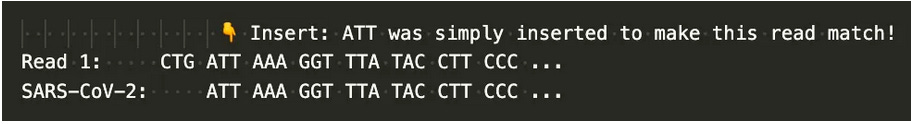
Οι αναντιστοιχίες είναι όταν η βάση δεν ταιριάζει με τη βάση που βρίσκεται στην ίδια θέση στο γονιδίωμα αναφοράς. Τα ένθετα είναι βάσεις που παρεμβάλλονται, οι οποίες προστίθενται, προκειμένου η ανάγνωση να ευθυγραμμιστεί σωστά.
Ακολουθεί ένα παράδειγμα, με βάση την πρώτη ανάγνωση, εδώ έχουν απλώς εισαγάγει το "ATT" στη θέση 4, το οποίο επισημαίνεται επίσης με το μοβ εικονίδιο στο παραπάνω στιγμιότυπο οθόνης.
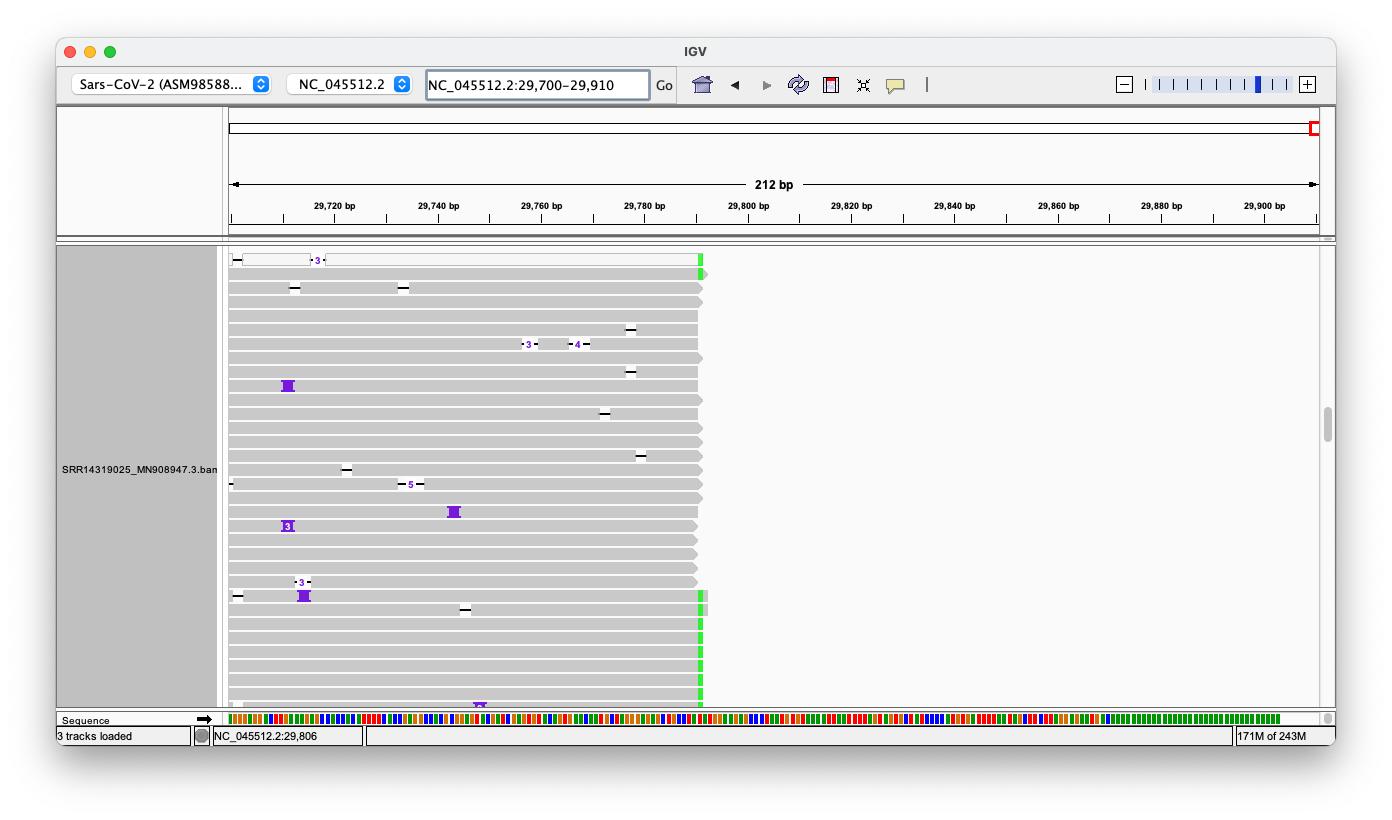
Ουρά
Η ουρά φαίνεται ακόμη χειρότερη, καθώς καμμία από τις αναγνώσεις δεν ταυτίζεται καν πλήρως με το τέλος. Υπήρχαν μόνο δύο αναγνώσεις που πλησίασαν κάπως, αλλά εξακολουθούσαν να έχουν 4-6 σφάλματα ανάγνωσης σε λιγότερο από 100 βάσεις. Το σύντομο διαφανές/λευκό ανάγνωσμα επισημαίνεται στην πραγματικότητα ως χαμηλής ποιότητας από το IGV. Έχω επισημάνει τις αναντιστοιχίες με μοβ κύκλο.
Συζήτηση
Καμμία από τις αναγνώσεις δεν ευθυγραμμίζεται τέλεια, ούτε με την κεφαλή ούτε με την ουρά. Ως εκ τούτου, μου είναι ασαφές πώς αυτή η αλληλουχία θα μπορούσε να έχει καθιερωθεί ως το γονιδίωμα αναφοράς για τον SARS-CoV-2. Για να διαπιστωθεί ότι πρόκειται πράγματι για το γονιδίωμα αναφοράς, θα πρέπει τουλάχιστον να αποδειχθεί ότι:
Τουλάχιστον 10-20 αναγνώσεις που παρουσιάζουν τέλεια ευθυγράμμιση με την κεφαλή και την ουρά.
Στο δείγμα υπάρχει πλήρης γενετική αλληλουχία (RNA) μήκους ~30kb.
Εξ όσων γνωρίζω, καμμία από αυτές τις δύο προϋποθέσεις δεν έχει εκπληρωθεί μέχρι σήμερα.
Ποιότητα περικομμένων αναγνώσεων
Στην παραπάνω επεξήγηση, χρησιμοποίησα τα reads όπως παρέχονται από τους Wu et al. 2020. Δεν είναι σαφές για μένα, αν αυτά είναι ήδη ποιοτικά περικομμένα. Έτσι, χρησιμοποίησα το πρόγραμμα fastp και trimmomatic για να κάνω πρώτα ποιοτική περικοπή και στη συνέχεια ευθυγράμμιση, αλλά αυτό δεν οδήγησε σε σημαντικά καλύτερα ή πειστικά αποτελέσματα. Παρόλα αυτά, παραμένει σημαντικό να κατανοήσουμε πώς το πρώτο γονιδίωμα θα μπορούσε να έχει καθοριστεί με επιστημονική βεβαιότητα, γι' αυτό και η εστίαση στο σύνολο δεδομένων των Wu et al. 2020 είναι τόσο σημαντική!
Τεχνολογίες αλληλούχισης πλήρους μήκους
Ενώ η εστίαση στην αρχική μελέτη Wu et al. 2020 που καθορίζει το γονιδίωμα αναφοράς είναι πιο σημαντική - ένα κοινό επιχείρημα για την αξιοπιστία της βαθιάς μεταγονιδιωματικής αλληλούχισης, είναι ότι υπάρχουν άλλες τεχνολογίες, που μπορούν να αλληλουχήσουν μεγαλύτερες αναγνώσεις (1-2kb) ή ενδεχομένως ακόμη και το πλήρες γονιδίωμα.
Oxford Nanopore
Ως εκ τούτου, το επανέλαβα και με ένα Nanopore SRR, χωρίς αξιοσημείωτες διαφορές στο αποτέλεσμα.
Σχεδόν σε όλες τις αναγνώσεις φαίνεται να υπάρχουν σημαντικά προβλήματα με την ευθυγράμμιση, που οδηγούν σε αναντιστοιχίες, εισαγωγές ή διαγραφές.
Δεν μπόρεσα να βρω ακατέργαστες αναγνώσεις για αλληλούχιση μακράς ανάγνωσης Nanopore, που να περιλαμβάνουν και τα άκρα. Αυτή η έκδοση, χρησιμοποιεί το πρωτόκολλο Midnight v3, το οποίο παραλείπει συγκεκριμένα τα άκρα.
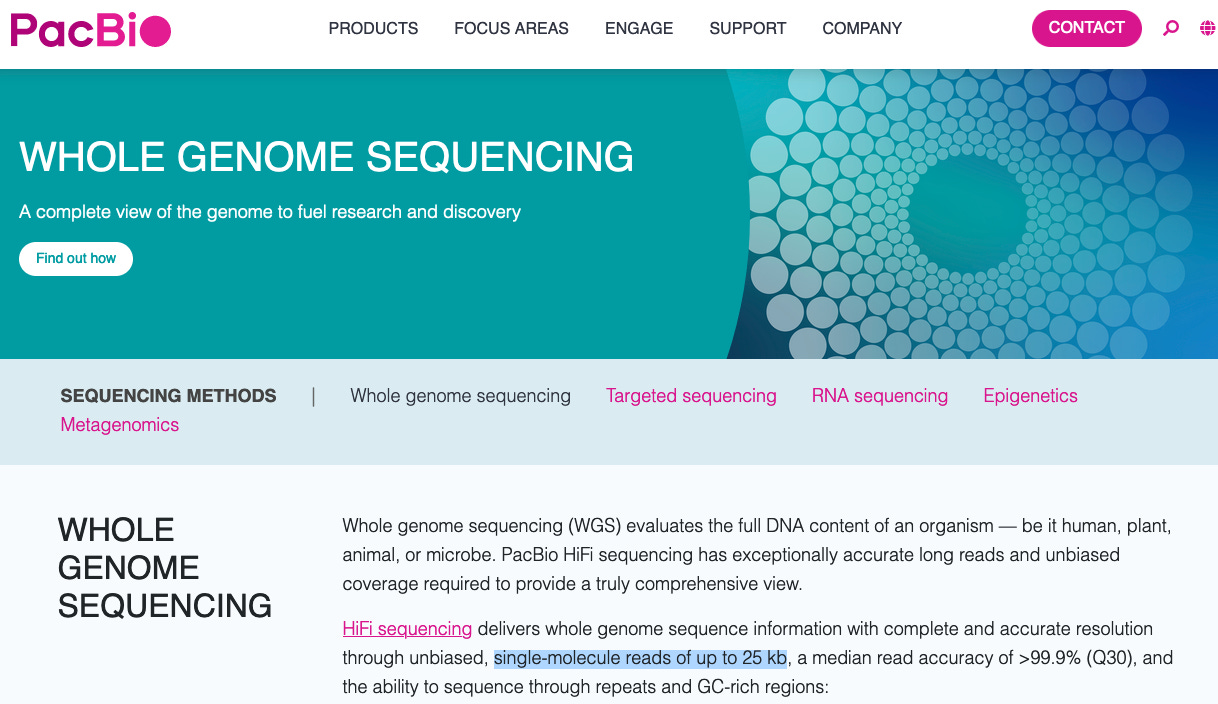
PacBio
Η εταιρεία PacBio ισχυρίζεται, στον ιστότοπό της, ότι μπορεί να προβεί σε αλληλουχία "αναγνώσεων ενός μορίου έως και 25 kb".
Παρέχουν δεδομένα δείγματος από την "αλληλούχιση HIFI" του SARS-CoV-2 - αλλά όταν το εξετάζουμε στο IGV, η ουρά και η κεφαλή δεν ευθυγραμμίζονται καθόλου (λείπουν εντελώς).
Συμπέρασμα
Δεν μπόρεσα να βρω καμία πειστική απόδειξη ότι το γονιδίωμα αναφοράς SARS-CoV-2 είναι έγκυρο, ειδικά όταν εξετάζω τις ευθυγραμμίσεις κεφαλής και ουράς.
Πηγές και κώδικας
Μπορείτε να βρείτε όλο τον κώδικα και τα αποτελέσματα που αναφέρονται σε αυτό το άρθρο εδώ:
https://github.com/USMortality/Megahit-SARS-CoV-2
Δικτυογραφία:
Is the SARS-CoV-2 Genome Valid? - USMortality