Συναρμολόγηση του Γονιδιώματος (Genome Assembly) του SARS-CoV-2 -- [Μέρος 2ο]
Μια επικαιροποίηση της ανάρτησής μου πριν από έξι μήνες.
Μετάφραση: Απολλόδωρος
27 Μαρτίου 2023 | USMORTALITY | Διάβαστε το εδώ.
Ιστορικό
Πριν από περίπου έξι μήνες, έγραψα ένα άρθρο σχετικά με τη συναρμολόγηση του γονιδιώματος του SARS-CoV-2. Σε αυτό το άρθρο, δημοσίευσα ένα σενάριο που αποσκοπούσε στην αναπαραγωγή των ευρημάτων των Wu et al., 2020, τα οποία οδήγησαν στην πρώτη δημοσιευμένη γονιδιωματική αλληλουχία του ιού SARS-CoV-2, στην οποία βασίζεται ολόκληρη η πανδημία.
Τα δύο κύρια ευρήματά μου τότε ήταν τα εξής:
Υπάρχουν τρεις δημοσιευμένες εκδόσεις του γονιδιώματος, και καμμία από αυτές δεν ήταν ακριβώς αναπαραγώγιμη με την εκτέλεση του Megahit (του de-novo assembler που χρησιμοποιήθηκε για την παραγωγή του μακρύτερου contig που χρησιμοποιήθηκε για το γονιδίωμα).
Το πλήρες γονιδίωμα δεν ήταν αναπαραγώγιμο μόνο με το Megahit. Ακόμη και οι συγγραφείς δεν ισχυρίστηκαν ποτέ κάτι τέτοιο, καθώς είπαν ότι χρησιμοποίησαν το RACE για να προσδιορίσουν "τα πλήρη τερματικά του γονιδιώματος".
Για το πλαίσιο της έρευνας: "Η RACE μπορεί να παρέχει την αλληλουχία ενός μεταγράφου RNA από μια μικρή γνωστή αλληλουχία εντός του μεταγράφου έως το 5' άκρο (5' RACE-PCR) ή το 3' άκρο (3' RACE-PCR) του RNA". Ουσιαστικά, μια μικρή περιοχή του τέλους του contig χρησιμοποιείται για έναν εκκινητή PCR, για να βρεθεί δήθεν το τέλος του γονιδιώματος. Πώς είναι δυνατόν, να γνωρίζουμε πού πραγματικά είναι το τέλος και αν ο συναρμολογητής έχει πραγματικά σταματήσει τη συναρμολόγηση κοντά στο τέλος, παραμένει ασαφές.
Ενώ κατάφερα να συναρμολογήσω τον πυρήνα του γονιδιώματος, τα παραγόμενα contigs διέφεραν σημαντικά σε μήκος από αυτό που είχαν δημοσιεύσει οι Wu et al. Στα σχόλια του άρθρου μου, ορισμένοι επεσήμαναν ότι μου έλειπε ένα βήμα από τον αγωγό μου: "Οι αναγνώσεις αλληλούχισης πρώτα προσαρμόστηκαν και περικόπηκαν ποιοτικά χρησιμοποιώντας το πρόγραμμα Trimmomatic".
Εκτός από πολλά από τα ανοιχτά ερωτήματα που έθεσα στο προηγούμενο άρθρο μου, τα λίγα βασικά ζητήματα με τη δημοσίευση των Wu et al. 2020 είναι τα εξής:
Δεν είναι σε θέση να αναπαράγουν τα 384.096 contigs με βάση τα δεδομένα προέλευσης, (μόνο 22.566 contigs).
Υπάρχουν τρεις εκδόσεις του ισχυριζόμενου γονιδιώματος που έχουν αναρτηθεί στη Genbank
Το μεγαλύτερο contig ισχυρίζεται ότι έχει: "30.474 νουκλεοτίδια (nt)", αλλά οι τρεις εκδόσεις του MN908947 αναφέρουν μόνο ένα contig με 30.473 ζεύγη βάσεων.
Μέθοδοι & Αποτελέσματα
Καθώς το βήμα της ποιοτικής περικοπής έλειπε νωρίτερα, ενημέρωσα τώρα το σενάριο και έκανα εκ νέου τη συναρμολόγηση. Ακολουθούν τα αποτελέσματα, μαζί με μερικές ακόμη πληροφορίες και την κατανόηση που συγκέντρωσα στην πορεία.
Το πρόσθετο βήμα περικοπής έδωσε αυτό το αποτέλεσμα:
Τώρα, λοιπόν, που έχουμε "περικόψει" ποιοτικά τις αναγνώσεις, ας τρέξουμε ξανά το Megahit. Όπως σημειώθηκε στο προηγούμενο άρθρο, ο Wu έτρεξε το Megahit με την έκδοση 1.1.3, ενώ η τελευταία έκδοση ακόμη και τότε ήταν ήδη η 1.2.9 - συνεπώς ακολουθούν τα αποτελέσματα και για τις δύο εκδόσεις.
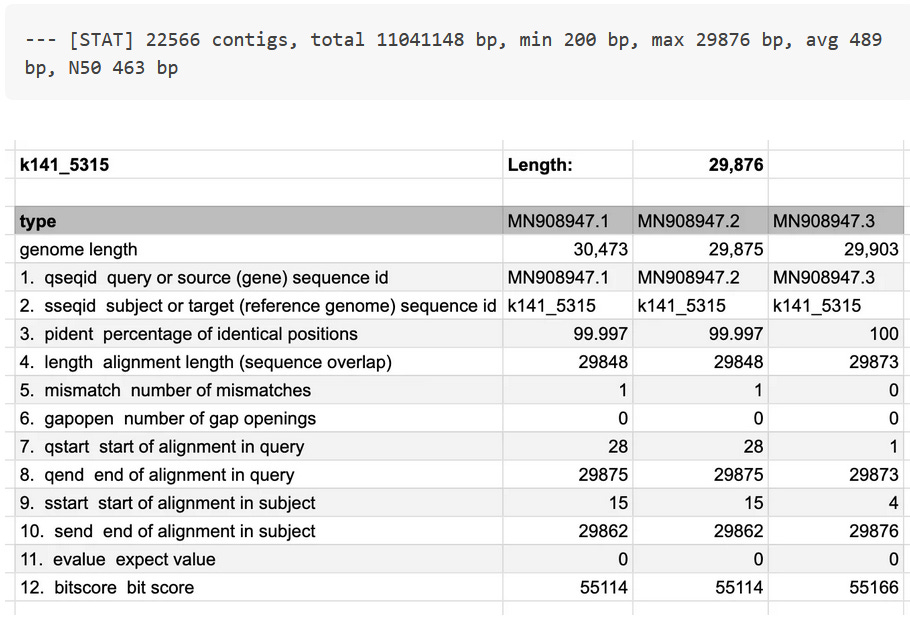
Megahit 1.2.9
Με την τρέχουσα έκδοση του Megahit, συναρμολογεί το μεγαλύτερο contig με 29.875 ζεύγη βάσεων.
Ωστόσο, συγκρίνοντας αυτό το contig με τις τρεις εκδόσεις του γονιδιώματος του Wu, δεν έχουμε ούτε τέλεια ταύτιση. Η πρώτη έκδοση έχει πολύ περισσότερα ζεύγη βάσεων και υπάρχει μία αναντιστοιχία. Η δεύτερη έκδοση ταιριάζει με το συνολικό μήκος, αλλά μόνο 29.848 ζεύγη βάσεων ταιριάζουν, εκτός από μία αναντιστοιχία. Η τρίτη εκδοχή είναι πιθανώς η πιο κοντινή, καθώς δεν υπάρχει πλέον καμία αναντιστοιχία, άρα 100% ίδιες θέσεις για 29.873 ζεύγη βάσεων, αλλά το συνολικό μήκος είναι εκτός.
Κατά την προσεκτικότερη εξέταση της διαφοράς με το τρίτο γονιδίωμα, μπορούμε να δούμε ότι μόνο τα δύο πρώτα γράμματα είναι διαφορετικά και ότι λείπει η ουρά (η οποία πιθανότατα προστέθηκε χειροκίνητα):
Megahit 1.1.3
Η έκδοση που οι Wu et al. τεκμηριωμένα χρησιμοποίησαν, έδωσε αυτό το αποτέλεσμα, το οποίο δεν πλησίασε ούτε καν το αρχικώς υποστηριζόμενο μεγαλύτερο περίγραμμα των 30.474 bp:
Συγκρίνοντας ξανά αυτό το contig με τις τρεις εκδόσεις του γονιδιώματος του Wu. Η πρώτη έκδοση έχει πολύ περισσότερα ζεύγη βάσεων, υπάρχει μία αναντιστοιχία. Η δεύτερη έκδοση έχει συνολικά ένα ζεύγος βάσεων λιγότερο, αλλά μόνο 29.848 ζεύγη βάσεων ταιριάζουν, συμπεριλαμβανομένου 1 αναντιστοιχίας. Η τρίτη εκδοχή κατά είναι η πιο κοντινή, καθώς δεν υπάρχει πλέον καμιά αναντιστοιχία, άρα 100% ταυτόσημες θέσεις για 29.873 ζεύγη βάσεων, αλλά το συνολικό μήκος είναι και πάλι εκτός.
Ας επιθεωρήσουμε και πάλι τις διαφορές με την τρίτη έκδοση, και έχουμε μια παρόμοια εικόνα όπως πριν, μόνο που αυτή τη φορά τα τρία πρώτα ζεύγη βάσεων είναι διαφορετικά, και η ουρά λείπει πάλι.
Συζήτηση
Έχω δείξει ότι, ακόμη και με το προηγούμενο βήμα της ποιοτικής περικοπής των αναγνώσεων, δεν μπορώ να αναπαράγω τα ακριβή αποτελέσματα που κατέγραψαν οι Wu et al 2020 στην εργασία τους. Μέχρι σήμερα, και εξ όσων γνωρίζω, κανείς δεν έχει καταφέρει να αναπαραγάγει το μακρύτερο υποστηριζόμενο 30.474 contig από τα παρεχόμενα ακατέργαστα δεδομένα.
Επιπλέον, ένας σχολιαστής του τελευταίου μου άρθρου ανέφερε ότι ο Wu μπορεί να είχε εκτελέσει την αρχική συναρμολόγηση στις μη φιλτραρισμένες αναγνώσεις (για την αντιστοίχιση με το ανθρώπινο γονιδίωμα), οι οποίες δυστυχώς δεν δημοσιεύονται για λόγους πλήρους διαφάνειας. Ο σχολιαστής ανέφερε επίσης, ότι ακόμη και οι φιλτραρισμένες αναγνώσεις, μετά την τελευταία ενημέρωση του ανθρώπινου γονιδιώματος, αποδίδουν μερικές ακόμη αντιστοιχίες, οι οποίες προηγουμένως δεν θα είχαν φιλτραριστεί. Αν και δεν έχω προσπαθήσει προσωπικά να το επαληθεύσω αυτό ακόμη - αν αληθεύει - αυτό θα ήταν ανησυχητικό, διότι δεν γνωρίζουμε τι θα αποκαλύψουν οι μελλοντικές ενημερώσεις του ανθρώπινου γονιδιώματος και αν οι αναγνώσεις είναι πράγματι μη ανθρώπινης προέλευσης.
Παρόλο που είναι δυνατόν να αναπαραχθεί σχεδόν ολόκληρο το ισχυριζόμενο γονιδίωμα με το Megahit, εξακολουθεί να είναι σαφές, ότι η ουρά πάντα λείπει, και άλλοι συναρμολογητές έχουν αποτύχει να πλησιάσουν έστω και στο ελάχιστο αυτά τα αποτελέσματα. Αυτό έχει επίσης επιβεβαιωθεί ως τέτοιο από τους Islam, et al. 2021, οι οποίοι δηλώνουν, ότι "[...] ολόκληρο το ιϊκό γονιδίωμα δεν συναρμολογήθηκε στις περισσότερες περιπτώσεις, ειδικά στις άκρες του γονιδιώματος".
Συνεχίζουν, για να ζητήσουν καλύτερους συναρμολογητές, και στη συνέχεια αναφέρουν αυτή την κυκλική πλάνη: "Το γονιδίωμα του ιού SARS-CoV-2 θα μπορούσε επίσης να συναρμολογηθεί με την ευθυγράμμιση των αναγνώσεων με το γονιδίωμα αναφοράς ή με τη χρήση μιας κατευθυνόμενης συναρμολόγησης αναφοράς". Η ευθυγράμμιση των αναγνώσεων, σημαίνει, να ευθυγραμμιστούν οι αναγνώσεις εισόδου ξανά με το συναρμολογημένο μακρύτερο contig (ή το τελικό γονιδίωμα, συμπεριλαμβανομένων των τερματικών). Προφανώς πρόκειται για πλάνη, καθώς το γονιδίωμα αναφοράς (αυτό που συζητήσαμε εδώ, από τους Wu et al. 2020) συναρμολογείται με την ίδια ακριβώς μέθοδο (de-novo assembly, Megahit) που απέτυχε να συναρμολογήσει ολόκληρο το γονιδίωμα εξ αρχής.
Μια άλλη ενδιαφέρουσα πτυχή είναι το ζήτημα της απομόνωσης, το οποίο φαίνεται να είναι πολύ αμφιλεγόμενο και μου έχει φέρει πολλές συζητήσεις στο Twitter. Από την εργασία των Wu et al. γίνεται σαφές, για τον άγρυπνο αναγνώστη, ότι οι Wu et al. σαφώς δεν είχαν ένα καθαρισμένο απομονωμένο δείγμα, διαφορετικά δεν θα υπήρχε ανάγκη να φιλτράρουν προγραμματισμένα τις αναγνώσεις που ταίριαζαν με το ανθρώπινο γονιδίωμα.
Ανοιχτά ερωτήματα
Τέλος, στο τελευταίο μου άρθρο έθεσα επίσης πολλά σημαντικά ερωτήματα, τα οποία δεν απαντήθηκαν επαρκώς από κανέναν μέχρι σήμερα, και τα οποία έχω τροποποιήσει με μερικά επιπλέον:
Γιατί το διεκδικούμενο γονιδίωμα ενημερώθηκε τόσες πολλές φορές (τρεις φορές);
(α) Γιατί άλλαξε το μήκος του;
(β) Γιατί η δημοσίευση αναφέρει 30.474 nt, ενώ το μεγαλύτερο κατατεθειμένο contig ήταν μόνο 30.473 nt;
Γιατί ο ασθενής μετακινήθηκε από νοσοκομείο σε νοσοκομείο ενώ βρισκόταν στη ΜΕΘ σε αναπνευστήρα, και ποια ήταν η τελική του έκβαση;
Είναι βέβαιο ότι όλα όσα έχουν απομείνει στο σύνολο των δεδομένων είναι μη ανθρώπινης προέλευσης;
Γιατί οι Wu et al. χρησιμοποίησαν την πολύ παλαιότερη έκδοση 1.1.3 του Megahit, αντί για την τελευταία έκδοση 1.2.9;
Γιατί το Trinity και το Megahit παρήγαγαν τόσο πολύ διαφορετικά αποτελέσματα;
Γιατί το μεγαλύτερο αρχικό contig (30.474nt) δεν μπορεί να συναρμολογηθεί με το Megahit;
Γιατί κανένας άλλος de-novo assembler (π.χ. trinity, spades) δεν είναι ούτε κατά διάνοια σε θέση να συναρμολογήσει το πλήρες γονιδίωμα;
Γιατί το πλήρες/ολόκληρο γονιδίωμα δεν έχει επικυρωθεί έναντι του αρχικού δείγματος ασθενούς (ή άλλων δειγμάτων);
Γιατί επιλέχθηκε το μακρύτερο contig, τι γίνεται με τα άλλα contigs από τα πάνω από 384 χιλιάδες συναρμολογημένα contigs;
Γιατί το Megahit συναρμολογεί μόνο περίπου 22 χιλιάδες contigs, όταν οι Wu et al ισχυρίζονται 384 χιλιάδες;
Πώς διαπιστώνεται, με βεβαιότητα, ότι το contig είναι ιικής προέλευσης;
Γιατί δεν δημοσιεύονται οι μη φιλτραρισμένες αναγνώσεις;
Γιατί το Megahit δεν είναι σε θέση να συναρμολογήσει την ουρά;
Πώς μπορεί να αποκλειστεί ότι κανένα άλλο μη εντοπισμένο παθογόνο δεν ευθύνεται για την ασθένεια (από τα άλλα 1,7 εκατ. contigs);
Πώς είναι δυνατόν να γνωρίζουμε πού βρίσκεται το πραγματικό τέλος του γονιδιώματος και αν ο συναρμολογητής σταμάτησε πραγματικά τη συναρμολόγηση κοντά στο τέλος;
Περίληψη
Συνοψίζοντας λοιπόν, μπορούμε να συμπεράνουμε ότι ούτε τα αποτελέσματα της δημοσίευσης των Wu et al. 2020, είναι ακριβώς αναπαραγώγιμα μέχρι σήμερα, ούτε το γονιδίωμα του SARS-CoV-2 έχει συναρμολογηθεί πλήρως με μία μόνο μέθοδο και/ή με έναν μόνο συναρμολογητή. Επιπλέον, παραμένουν πολλά σχετικά αναπάντητα ερωτήματα, τα οποία θα πρέπει να απαντηθούν από τους αρμόδιους υπεύθυνους.
Ένα μεγάλο ευχαριστώ στους αναγνώστες Mongol και Paul για τις γόνιμες συνεχείς συζητήσεις τους.
Περισσότερες πληροφορίες
Ο συναρμολογητής Megahit, που αναφέρεται σε αυτό το άρθρο, χρησιμοποιεί την προσέγγιση "de-Bruijn graph" για την de-novo συναρμολόγηση, για να καταλάβετε γρήγορα πώς αυτό λειτουργεί στην πράξη, δείτε αυτό το εξαιρετικό σύντομο βίντεο:
Πηγές & Κώδικας
Μπορείτε να βρείτε όλο τον κώδικα και τα αποτελέσματα που αναφέρονται σε αυτό το άρθρο εδώ:
Περαιτέρω πηγές:
https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&page_size=10&acc=SRR10971381&display=reads
https://www.metagenomics.wiki/tools/blast/blastn-output-format-6
Δικτυογραφία:
SARS-CoV-2 Genome Assembly (Part 2) - USMortality