Συναρμολόγηση του Γονιδιώματος του SARS-CoV-2
Προσπάθεια αναδημιουργίας της φερόμενης ως γενετικής αλληλουχίας του SARS-CoV-2.
Σας ευχαριστώ θερμά για το ενδιαφέρον σας και την αναδημοσίευση των άρθρων μου. Θα εκτιμούσα ιδιαίτερα αν, κατά την κοινοποίηση, σ̲υ̲μ̲π̲ε̲ρ̲ι̲λ̲α̲μ̲β̲ά̲ν̲α̲τ̲ε̲ ̲κ̲α̲ι̲ ̲τ̲ο̲ν̲ ̲σ̲ύ̲ν̲δ̲ε̲σ̲μ̲ο̲ ̲(̲l̲i̲n̲k̲)̲ ̲τ̲ο̲υ̲ ̲ά̲ρ̲θ̲ρ̲ο̲υ̲ ̲μ̲ο̲υ̲. Αυτό όχι μόνο αναγνωρίζει την πηγή, αλλά επιτρέπει και σε άλλους να ανακαλύψουν περισσότερο περιεχόμενο. Η υποστήριξή σας είναι πολύτιμη για τη συνέχιση της δουλειάς μου.
Απόδοση στα ελληνικά: Απολλόδωρος - US Mortality | 3 Οκτωβρίου 2022
Μπορείτε να κάνετε εφάπαξ ή επαναλαμβανόμενες δωρεές μέσω του Ko-Fi:
Εδώ προσπαθώ να αναπαραγάγω τον τρόπο με τον οποίο οι πρώτοι επιστήμονες αναγνώρισαν και αλληλούχισαν τον κορονοϊό SARS-CoV-2. Αυτό είναι το πρώτο άρθρο μιας σειράς τριών άρθρων, οπότε παρακαλούμε μείνετε συντονισμένοι, μοιραστείτε και εγγραφείτε!
Επισκόπηση της επταμερούς σειράς μου για τη γονιδιωματική:
Ιστορικό
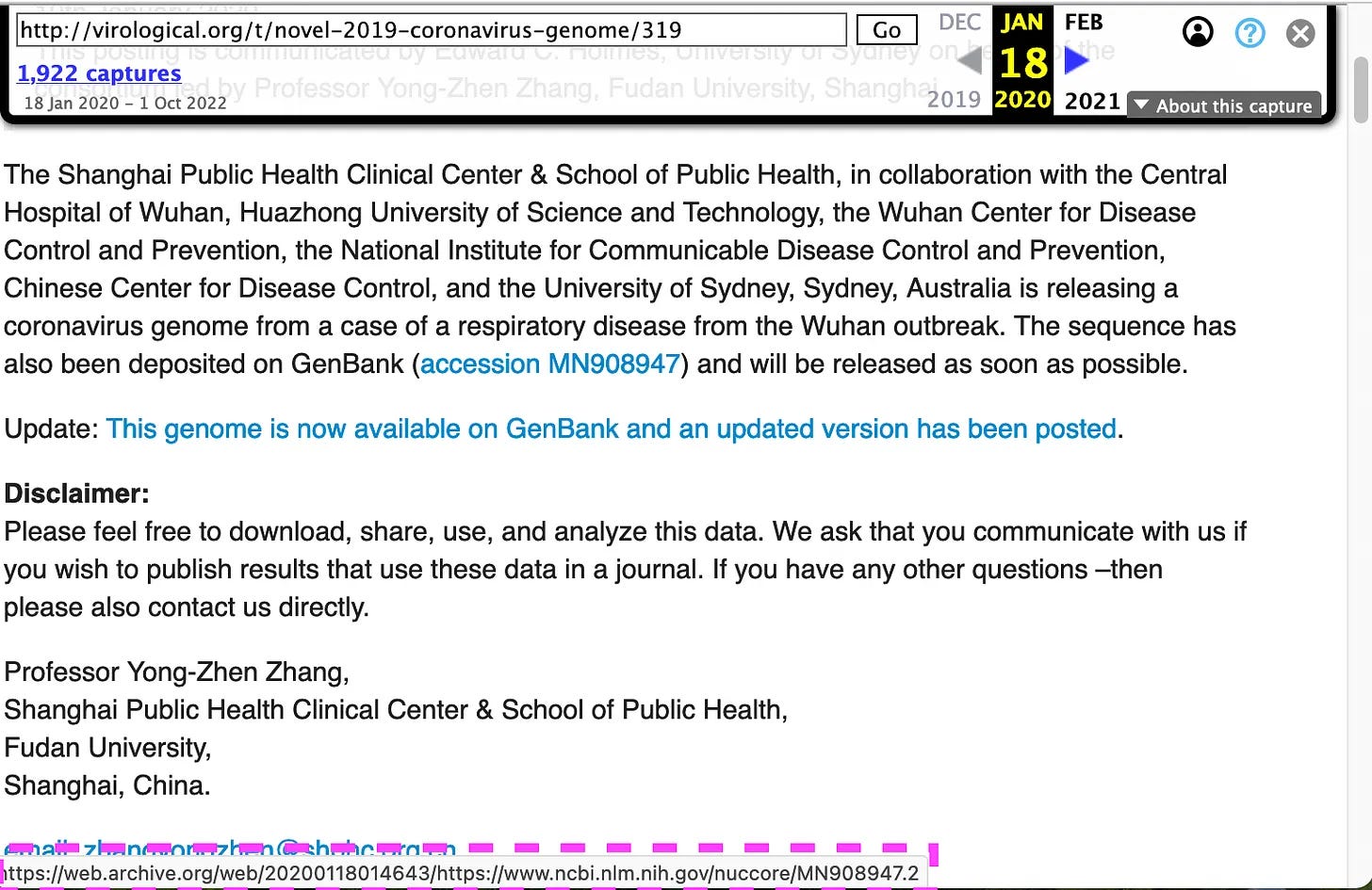
Το πρώτο σημείωμα σχετικά με τον νέο ιό δημοσιεύθηκε από τον καθηγητή Zhang (Κίνα) μέσω του λογαριασμού στο φόρουμ του καθηγητή Holmes (Αυστραλία) στις 20 Ιανουαρίου 2020.
https://virological.org/t/novel-2019-coronavirus-genome/319
Ερώτηση: Γιατί ο καθηγητής Zhang χρησιμοποίησε τον λογαριασμό του καθηγητή Holmes - ή γιατί ο τελευταίος δημοσίευσε τη δήλωση του φορμαλιστή;
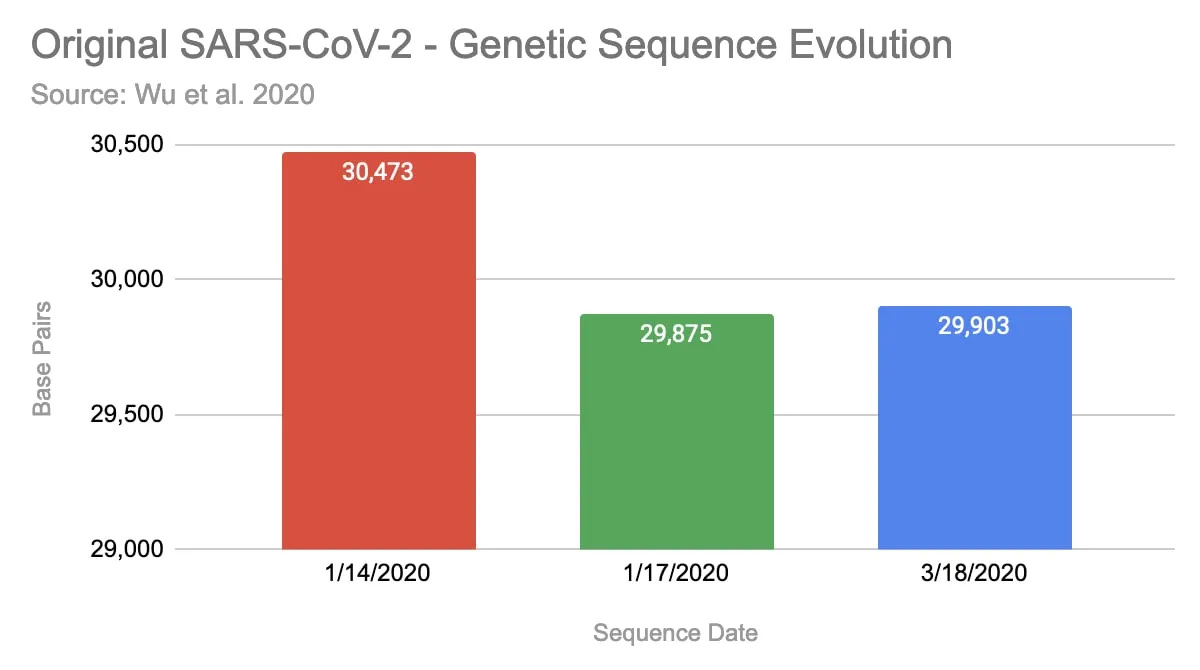
Αξίζει να σημειωθεί, ότι έχουν ανεβάσει τρεις εκδόσεις της ισχυριζόμενης ακολουθίας μέχρι τώρα. Η πρώτη έκδοση, που ανέβηκε στις 14 Ιανουαρίου 2020, περιείχε 30.473 ζεύγη βάσεων (bp). Τρεις ημέρες αργότερα την ενημέρωσαν με μια μικρότερη έκδοση με 29.875 bp και στις 18/3/2020 επεκτάθηκε και πάλι σε 29.903 bp.
https://www.ncbi.nlm.nih.gov/nuccore/MN908947
Η καταχώρηση στο φόρουμ που αναρτήθηκε στις 17/1 παραπέμπει στη δεύτερη έκδοση:
https://web.archive.org/web/20200118014643/http://virological.org/t/novel-2019-coronavirus-genome/319
Μέχρι στιγμής, δεν έχω βρει καμία πληροφορία γιατί το γονιδίωμα έχει ενημερωθεί τουλάχιστον τρεις φορές.
Ερ: Γιατί το γονιδίωμα ενημερώθηκε τόσες πολλές φορές;
Οι Zhang & Wu δημοσίευσαν το συνοδευτικό έγγραφο για την ακολουθία εδώ:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7094943/
Εδώ περιγράφουν πώς πήραν δείγμα από έναν «41χρονο άνδρα», με τα ακόλουθα συμπτώματα: «πυρετός, σφίξιμο στο στήθος, μη παραγωγικός βήχας, πόνος και αδυναμία για 1 εβδομάδα». Του χορηγήθηκαν διάφορα φάρμακα, αλλά παρόλα αυτά χρειάστηκε να τεθεί σε αναπνευστήρα στη ΜΕΘ. Αναφέρουν ότι αργότερα μεταφέρθηκε σε άλλο νοσοκομείο - κάτι που είναι παράλογο, δεν είμαι σίγουρος γιατί άνθρωποι που βρίσκονται σε αναπνευστήρα και σε ΜΕΘ μετακινούνται μεταξύ νοσοκομείων. Η έκβαση του ασθενούς παραμένει άγνωστη από την εφημερίδα.
Ερ: Γιατί ο ασθενής μετακινήθηκε μεταξύ νοσοκομείων ενώ βρισκόταν σε ΜΕΘ σε αναπνευστήρα και ποια ήταν η τελική έκβαση;
Αλληλουχία
Στην εργασία, οι συγγραφείς περιγράφουν πώς δημιούργησαν την αλληλουχία.
Πήραν λοιπόν βρογχικό υγρό από αυτόν τον ασθενή, το μετέτρεψαν σε DNA και το επεξεργάστηκαν με ένα Sequencer, το οποίο διαβάζει τον γενετικό κώδικα του μίγματος.
Αξίζει να σημειωθεί, ότι μόνο το DNA μπορεί να αλληλουχηθεί, επομένως έπρεπε πρώτα να μετατρέψουν όλο το RNA του δείγματος σε DNA. Αξίζει να σημειωθεί, ότι τα δικά μας κύτταρα, εσωτερικά, χρησιμοποιούν RNA και φυσικά αποτελούνται από DNA. Έτσι, μετά τη μετατροπή έχουν μόνο DNA το οποίο προέρχεται από ανθρώπινο DNA, ανθρώπινο RNA και ενδεχομένως RNA πολλών μικροβίων (συμπεριλαμβανομένων των ιών). Τώρα σε ένα τελευταίο βήμα γράφουν, ότι απέκλεισαν το ανθρώπινο DNA με την αντιστοίχισή του με το Ανθρώπινο Γονιδίωμα - αλλά:
Είναι βέβαιο ότι όλα όσα έχουν απομείνει στο σύνολο δεδομένων είναι μη ανθρώπινης προέλευσης;
Τώρα, το μηχάνημα διαβάζει μόνο αλληλουχίες μέχρι ένα μήκος 150bp, πράγμα που σημαίνει ότι δεν θα μπορούσε ποτέ να διαβάσει την πλήρη αλληλουχία οποιουδήποτε ιού. Αντ' αυτού, λαμβάνουμε ένα μεγάλο σύνολο των λεγόμενων αναγνώσεων με μήκος έως 150bps.
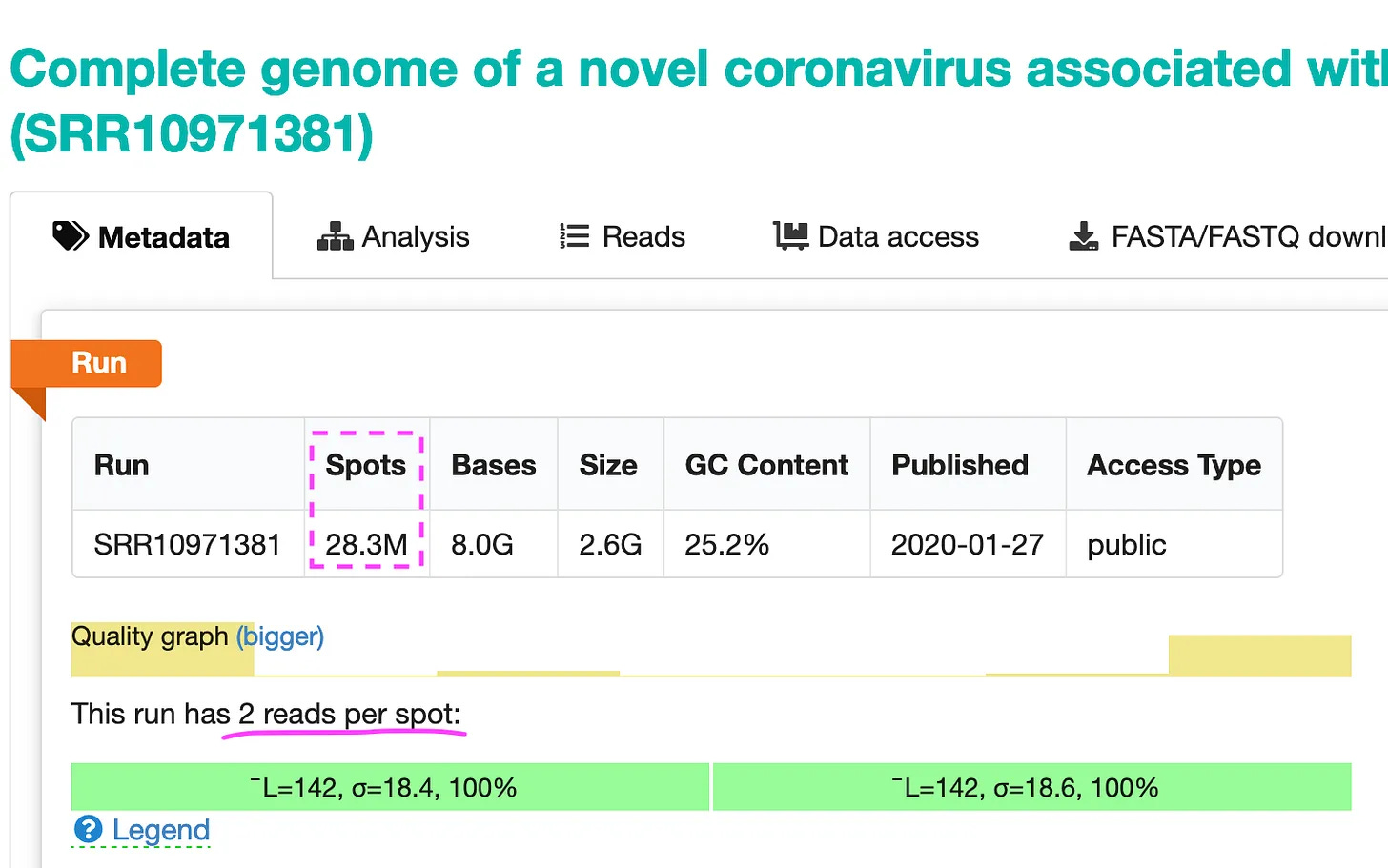
Έτσι, ο ακολουθιογράφος (sequencer) παρήγαγε 28,3 εκατομμύρια σημεία (με 2 αναγνώσεις ανά σημείο). Μοιάζουν όπως φαίνονται στην παρακάτω εικόνα.
Μπορείτε να ρίξετε μια ματιά σε όλες τις αναγνώσεις εδώ: https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&page_size=10&acc=SRR10971381&display=reads
Συναρμολόγηση
Τώρα που ξέρουμε από πού προήλθαν αυτές οι αναγνώσεις, ας προσπαθήσουμε να τις συναρμολογήσουμε με τα βιοπληροφορικά εργαλεία. Προηγουμένως έψαξα στο GitHub για ένα σενάριο που θα εκτελούσε απλά ολόκληρο το pipeline συναρμολόγησης, αλλά δεν κατάφερα να βρω κάποιο.
Έτσι δημιούργησα το δικό μου, και αυτό είναι το αποτέλεσμα:
Το σενάριο είναι διαθέσιμο εδώ: https://github.com/usmortality/Megahit-SARS-CoV-2
Η συναρμολόγηση γίνεται με ένα εργαλείο που ονομάζεται Megahit. Εγκαθίστανται όλες οι απαραίτητες εξαρτήσεις και τα «reads» κατεβαίνουν, μετατρέπονται σε μορφή fastq και στη συνέχεια εισάγονται στο Megahit. Τέλος, το αποτέλεσμα συμπιέζεται σε zip.
Έχω τρέξει αυτό το σενάριο σε μια βελτιστοποιημένη υπολογιστική περίπτωση 32 πυρήνων AWS ως εξής:
aws ec2 run-instances --image-id ami-026b57f3c383c2eec --count 1 --instance-type c5a.8xlarge --key-name ben --ebs-optimized --block-device-mapping «[ { \»DeviceName\«: \»/dev/xvda\«, \»Ebs\«: { \»VolumeSize\": 100 } } ]»scp megahit.sh megahit:/home/ec2-user/. ssh megahit chmod +x megahit.shοθόνη./megahit.shΑποτέλεσμα
Εδώ είναι το αποτέλεσμα της εφαρμογής μου με το Megahit 1.1.3
62,126Gb μνήμης συνολικά.Χρήση: Με τη χρήση: 55,913Gb.MEGAHIT v1.1.3--- [Sat Oct 1 16:42:12 2022] Έναρξη συναρμολόγησης. Αριθμός νημάτων CPU 32 ------ [Σαβ Οκτ 1 16:42:12 2022] Διαθέσιμη μνήμη: 66706915328, χρησιμοποιείται: 60036223795--- [Σαβ Οκτ 1 16:42:12 2022] Μετατροπή αναγνώσεων σε δυαδικά αρχεία ---b' [read_lib_functions-inl.h : 209] Lib 0 (SRR10971381_1.fastq,SRR10971381_2.fastq): pe, 56565928 reads, 151 max length'b' [utils.h : 126] Real: 46.1887\tuser: 35.0145\tsys: 10.3506\tmaxrss: 164224'--- [Sat Oct 1 16:42:58 2022] k list: 21,29,39,59,79,99,119,141 ------ [Σαβ Οκτ 1 16:42:58 2022] Εξαγωγή στερεών (k+1)-μερών για k = 21 ------ [Σαβ Οκτ 1 16:44:04 2022] Κατασκευή γραφήματος για k = 21 ------ [Sat Oct 1 16:44:55 2022] Συναρμολόγηση contigs από SdBG για k = 21 ------ [Σαβ Οκτ 1 16:47:27 2022] Τοπική συναρμολόγηση k = 21 ------ [Sat Oct 1 16:47:57 2022] Εξαγωγή επαναληπτικών ακμών από k = 21 έως 29 ------ [Sat Oct 1 16:48:52 2022] Κατασκευή γραφήματος για k = 29 ------ [Sat Oct 1 16:49:08 2022] Συναρμολόγηση contigs από SdBG για k = 29 ------ [Σαβ Οκτ 1 16:50:17 2022] Τοπική συναρμολόγηση k = 29 ------ [Sat Oct 1 16:50:50 2022] Εξαγωγή επαναληπτικών ακμών από k = 29 έως 39 ------ [Sat Oct 1 16:51:44 2022] Κατασκευή γραφήματος για k = 39 ------ [Sat Oct 1 16:51:54 2022] Συναρμολόγηση contigs από SdBG για k = 39 ------ [Sat Oct 1 16:52:54 2022] Local assembling k = 39 ------ [Sat Oct 1 16:53:26 2022] Εξαγωγή επαναληπτικών ακμών από k = 39 έως 59 ------ [Sat Oct 1 16:54:04 2022] Κατασκευή γραφήματος για k = 59 ------ [Sat Oct 1 16:54:12 2022] Συναρμολόγηση contigs από SdBG για k = 59 ------ [Σαβ Οκτώβριος 1 16:54:47 2022] Τοπική συναρμολόγηση k = 59 ------ [Sat Oct 1 16:55:22 2022] Εξαγωγή επαναληπτικών ακμών από k = 59 έως 79 ------ [Sat Oct 1 16:55:45 2022] Κατασκευή γραφήματος για k = 79 ------ [Sat Oct 1 16:55:48 2022] Συναρμολόγηση contigs από SdBG για k = 79 ------ [Σαβ Οκτώβριος 1 16:56:04 2022] Τοπική συναρμολόγηση k = 79 ------ [Sat Oct 1 16:56:37 2022] Εξαγωγή επαναληπτικών ακμών από k = 79 έως 99 ------ [Sat Oct 1 16:56:54 2022] Κατασκευή γραφήματος για k = 99 ------ [Sat Oct 1 16:56:56 2022] Συναρμολόγηση contigs από SdBG για k = 99 ------ [Sat Oct 1 16:57:03 2022] Τοπική συναρμολόγηση k = 99 ------ [Sat Oct 1 16:57:29 2022] Εξαγωγή επαναληπτικών ακμών από k = 99 έως 119 ------ [Sat Oct 1 16:57:41 2022] Κατασκευή γραφήματος για k = 119 ------ [Sat Oct 1 16:57:42 2022] Συναρμολόγηση contigs από SdBG για k = 119 ------ [Σαβ Οκτ 1 16:57:46 2022] Τοπική συναρμολόγηση k = 119 ------ [Sat Oct 1 16:58:09 2022] Εξαγωγή επαναληπτικών ακμών από k = 119 έως 141 ------ [Σαβ Οκτώβριος 1 16:58:16 2022] Κατασκευή γραφήματος για k = 141 ------ [Sat Oct 1 16:58:17 2022] Συναρμολόγηση contigs από SdBG για k = 141 ------ [Sat Oct 1 16:58:20 2022] Συγχώνευση για την παραγωγή τελικών περιγραμμάτων ------ [STAT] 30425 contigs, total 14483601 bp, min 200 bp, max 29870 bp, avg 476 bp, N50 448 bp--- [Σαβ Οκτ 1 16:58:20 2022] ΟΛΑ ΤΕΛΕΙΩΘΗΚΑΝ. Χρόνος που παρήλθε: 968.493221 δευτερόλεπτα ---https://github.com/USMortality/Megahit-SARS-CoV-2/blob/megahit-1.1.3/out/output.txt
Αξίζει να σημειωθεί, ότι οι Wu et al. χρησιμοποίησαν μια παλαιότερη έκδοση, συγκεκριμένα την 1.1.3 του Megahit από τον Μάρτιο του 2018, ενώ η τελευταία έκδοση 1.2.9 ήταν ήδη διαθέσιμη από τον Οκτώβριο του 2019.
Γιατί οι Wu et al. χρησιμοποίησαν την πολύ παλαιότερη έκδοση 1.1.3 του Megahit, αντί για την πιο πρόσφατη έκδοση 1.2.9;
Ως προς το γιατί χρησιμοποιήθηκε η συγκεκριμένη έκδοση παραμένει ασαφές.
https://github.com/voutcn/megahit/tree/v1.2.9
Επιστρέφοντας στα αποτελέσματα - βλέπουμε ότι η συναρμολόγηση διήρκεσε περίπου 16 λεπτά σε μια βελτιστοποιημένη για υπολογιστές 32 πυρήνων Amazon Linux instance.
Ακολουθούν τα τελικά στατιστικά στοιχεία:
[STAT] 30425 contigs, total 14483601 bp, min 200 bp, max 29870 bp, avg 476 bp, N50 448 bpΟι συγγραφείς έγραψαν στην εργασία τους, ότι η συναρμολόγησή τους απέδωσε:
Το Megahit παρήγαγε συνολικά 384.096 συναρμολογημένα contigs (εύρος μεγέθους 200-30.474 nt)
Έτσι, ενώ οι συγγραφείς συναρμολόγησαν 384k contigs, εγώ μπόρεσα να συναρμολογήσω μόνο 30k contigs (Αυτό μπορεί να έχει να κάνει με το πρόσθετο φιλτράρισμα που έκαναν).
Καθώς τα μεγαλύτερα contigs που δημιουργήθηκαν από το Megahit (30.474 nt)
Επίσης, δεν είμαι σε θέση να αναγεννήσω το μεγαλύτερο contig, το οποίο είχε πάνω από 30k bp, ενώ η δική μου εκτέλεση συναρμολόγησε μόνο 29,870bp.
Ακολουθούν τα αποτελέσματα συνοπτικά, τα δικά μου τρεξίματα είναι οι δύο μπάρες στα δεξιά:
Έτρεξα το Megahit 1.1.3 και 1.2.9, με το τελευταίο να παράγει ένα σημαντικά μικρότερο περίγραμμα με μόνο 29.802 bp. Τουλάχιστον, και οι δύο εκτελέσεις σε κάθε έκδοση παρήγαγαν το ίδιο μεγαλύτερο contig, εξαιρουμένων της αρχής και του τέλους.
Τέλος, είναι αξιοσημείωτο ότι καμία από τις ακολουθίες που συναρμολογήθηκαν από το Megahit δεν παρήγαγε το πλήρες διεκδικούμενο contig. Επίσης, το πλήρες γονιδίωμα δεν συναρμολογήθηκε ποτέ, καθώς το τέλος του πλήρους γονιδιώματος πάντα λείπει.
Περισσότερα για αυτό στο επόμενο άρθρο μου....
Παρακαλώ, ενημερώστε με για τις σκέψεις και τα σχόλιά σας στα σχόλια και μην ξεχάσετε να μοιραστείτε αυτό το άρθρο αν σας άρεσε!
Δείτε επίσης τον κώδικα: https://github.com/USMortality/Megahit-SARS-CoV-2
Δείτε επίσης το επόμενο άρθρο μου σχετικά με αυτό το θέμα:
Αν σας άρεσε αυτό το άρθρο, μοιραστείτε το, εγγραφείτε για να λαμβάνετε περισσότερο περιεχόμενο και αν θέλετε να στηρίξετε το συνεχές έργο μου, μπορείτε να χρησιμοποιήσετε τον παρακάτω σύνδεσμο.
—Δικτυογραφία:
SARS-CoV-2 Genome Assembly - by US Mortality - USMortality